Examensarbete i Biomedicinsk laboratorievetenskap Malmö Universitet
15 hp Hälsa och samhälle
Biomedicinsk analytikerprogrammet 205 06 Malmö Mars 2019
UTVÄRDERING AV
DNA-EXTRAKTIONSPROTOKOLL FÖR
FORENSISKA PROVER
2
UTVÄRDERING AV
DNA-EXTRAKTIONSPROTOKOLL FÖR
FORENSISKA PROVER
KASSEM AWAD
Awad K. Utvärdering av DNA-extraktionsprotokoll för forensiska prover.
Examensarbete i Biomedicinsk laboratorievetenskap 15 högskolepoäng. Malmö
universitet: Fakulteten för hälsa och samhälle, Institutionen för biomedicinsk laboratorievetenskap, 2019
Vid låga mängder av DNA, vilket det ofta är vid forensiska prover, är en lyckad DNA-analys beroende av ett effektivt DNA-extraktionsprotokoll. Bland flera kommersiellt tillgängliga extraktionsmetoder för DNA har tidigare studier visat att Chelex 100 resin, som idag används på Nationellt Forensiskt Centrum i
Linköping, är en snabb och enkel metod utan toxiska ämnen, långa väntetider eller större kostnader och som även ger ett högt DNA-utbyte. På teknisk mikrobiologi i Lund har olika chelex-baserade protokoll för spårsäkring och extraktion tagits fram för att optimera det aktuella protokollet på Nationellt Forensiskt Centrum. I detta arbete kombinerades spårsäkringsteknik och DNA-extraktionsprotokoll från tidigare studier med olika rör och tops, med syftet att i bästa fall generera ett protokoll som är optimalt för de vanligast förekommande brottsplatsproverna. I resultatet observerades högre DNA-utbyte med reducerade volymer
lyseringsbuffert, vilket användning av rör-i-rörsystem tillät. När rör-i-rörsystem sedan kombinerades med extraktionsprotokoll innehållande SDS, observerades en ökning i DNA-utbytet. När dessa två faktorer vidare kombinerades med en tops vars skaft kan klippas rakt av ökade DNA-utbytet ytterligare. Ett byte från mikrofugrör, som används på Nationellt Forensiskt Centrum idag, till ett rör-i-rörsystem reducerar lyseringsvolymen från ca. 600 µl till 200 µl vilket är en avgörande faktor för effektivare extraktioner.
Nyckelord: Autolys A rör, Chelex resin, Detergent, DNA-extraktion, qPCR, Saliv,
3
EVALUATION OF DNA EXTRACTION
PROTOCOLS FOR FORENSIC
SAMPLES
KASSEM AWAD
Awad K. Evaluation of DNA extraction protocols for forensic samples. Degree
project in biomedical science 15 credit points. Malmö University: Faculty of
Health and Society, Department of Biomedical Science, 2019
When handling low amounts of DNA, which often is the case with forensic samples, a successful DNA analysis is dependent on an effective DNA extraction protocol. Among many commercially available extraction methods for DNA, previous research has proved Chelex 100 resin, which is used at National Forensic Center in Linkoping, to be a quick and simple method without toxic substances, long waiting time or large costs, which in addition provides a high DNA
exchange. At Applied Microbiology, Lund´s University, different chelex-based protocols for sampling and DNA extraction have been developed to optimize the present protocol at the National Forensic Center. In this study, sampling
techniques and DNA extraction from previous studies was further combined with different tubes and swabs with the aim to, in best case, generate a protocol that is optimal for the most common crime scene samples. The results showed improved DNA yields with less extraction volume, which the tube in tube system made possible. When tube in tube system was combined with extraction protocols containing SDS, a higher DNA yield was observed. When these two factors were further combined with a cotton swab, whose shaft can be cut straight of, the DNA yield improved even more. A change from microfuge tubes, which is used at the National Forensic Center, to tube in tube system, the lysis volume could be
reduced from approximately 600 µl to 200 µl which is a deciding factor for higher DNA yield.
Keywords: Autolyse A tubes, Chelex resin, Detergent, DNA extraction, qPCR,
4
INNEHÅLL
BAKGRUND ... 5
DNA-profileringens utveckling ... 5
Den forensiska analyskedjan ... 5
Spårsäkring ... 5
DNA-extraktion ... 5
DNA-kvantifiering med kvantitativ PCR (qPCR) ... 6
DNA-profilering ... 7
Optimering av DNA-extraktionsmetoder ... 8
Syfte ... 8
Etisk bedömning ... 8
METOD OCH MATERIAL ... 9
Saliv som DNA-källa ... 9
Beredning och säkring av forensiska spår- Tops ... 9
DNA-extraktion ... 9
ChelexDirekt (NFC) extraktion ... 9
Extraktion med thermoskak ... 10
Autolysextraktion med TWEEN 20/SDS ... 10
Optimering av protokoll ... 10 Tops ... 10 Extraktionsrör ... 11 Undersökning av PCR-hämning ... 11 DNA-kvantifiering med qPCR ... 12 Försök på NFC ... 12 Iscensatt brottsplats ... 12 Analys av resultat ... 13 RESULTAT ... 13 Val av protokoll ... 13 Val av extraktionsrör ... 14 Undersökning av PCR-hämning ... 15 Val av tops... 15 Kombinationstester ... 16 Försök på NFC, Linköping ... 17
Utvärdering av klippning för Puritantops ... 19
Slutlig jämförelse av de mest lovande extraktionsprotokollen ... 20
Iscensatt brottsplats ... 20 DISKUSSION ... 21 Iscensatt brottsplats ... 23 Metod ... 23 PCR-inhibering ... 24 KONKLUSION ... 24 REFERENSER ... 26
5 Multiplex-PCR Kapillär-elektrofores Utvärdering av DNA-profil
BAKGRUND
DNA-profileringens utvecklingDet som idag benämns DNA-profilering har haft sin utveckling de senaste 75 åren genom en rad upptäckter. Redan 1944 beskrev Oswald Avery cellkomponenten deoxyribonukleinsyra (DNA), som en överföringsmekanism av ärftliga
karaktärsdrag. Ett decennium senare beskrev James Watson och Francis Crick DNA-molekylens struktur som en dubbelhelix. År 1980 blev David Botstein och hans kollegor först med att utnyttja små variationer mellan människor på genetisk nivå och konstruera en genetisk karta. Den genetiska variation som de använde sig av kallas restriction fragment length polymorphism (RFLP). 1984 upptäckte Alec Jeffrey en unik applikation för RFLP-tekniken; personlig identifikation. Tekniken, kom senare, med modifikation, att benämnas ”DNA fingerprinting” [1-2]. Detta introducerades kort därefter till kriminaltekniska laboratorium för
brottsutredningar i USA. ”DNA-fingerprinting” har med åren antagit en, vad som anses av flera forskare, mer passande term: DNA-profilering [1]. Då Kary Mullis 1986 uppfann metoden polymerase chain reaction (PCR) har denna blivit ett mycket viktigt och ovärderligt verktyg för DNA-analyser inom forensik. PCR har bidragit till en utveckling av snabbare och känsligare metoder med högre
urskiljning än vad som tidigare var möjligt [1,3]. Den forensiska analyskedjan
Idag består den forensiska analyskedjan för DNA-profilering av spårsäkring, DNA-extraktion, DNA-kvantifiering, multiplex PCR, kapillärelektrofores och utvärdering av DNA-profil (Figur 1).
Figur 1. Den forensiska analyskedjan
Spårsäkring
Det första steget i analyskedjan är spårsäkring, som kan utföras både på brottsplats och på forensiska laboratorium. Vid spårsäkring är det viktigt att maximera spårupptaget och minimera kontaminanter genom lämplig hantering och preservation av provet. Detta bör innefattas av skyddande klädsel, intelligent byte av handskar, rena och DNA-fria implement (t.ex. bomullstopsar, som är det vanligaste redskapet för svabbning idag) och passande förvaring av provet [1].
DNA-extraktion
Nästa steg i analyskedjan är att extrahera DNA:t och sedan kvantifiera mängden som extraherats. Vid låga mängder DNA, vilket är vanligt för forensiska prover, är en lyckad PCR-analys beroende av ett effektivt DNA-extraktionsprotokoll. Bland flera kommersiellt tillgängliga extraktionsmetoder för DNA är ”Chelex 100 resin” en snabb och enkel metod utan toxiska ämnen, långa väntetider eller större kostnader [4-6]. Dessutom sker hela extraktionen utan överföring mellan rör vilket annars skulle introducera risk för kontamination [5]. Chelex är ett chelerande resin innehållande styren divinylbensen copolymerer som består av parade iminodiacetatjoner. De parade iminodiacetatjonerna fungerar som
chelerande grupper för polyvalenta metalljoner som exempelvis magnesiumjoner
Spårsäkring DNA-extraktion
DNA-kvantifiering
6
[7]. När magnesiumjonerna, som är kritiska kofaktorer för deoxyribonukleaser (DNaser), inte är fria längre och därmed oanvändbara för DNaser, skyddas därmed DNA:t från att degraderas. Noterbart är att om Chelex förekommer i PCR-reaktionen kan det leda till polymerasinhibition eftersom DNA-polymeraser även utnyttjar Mg2+ som kofaktor [8]. Utöver att fungera som en kofaktor till DNA-polymeraset, ökar också Mg2+ affiniteten mellan primers och DNA genom att minska den elektrostatiska repulsionen mellan DNA:s fosfat ”backbone” [9].
Det finns många faktorer som påverkar effektiviteten i DNA-extraktionen och därmed DNA-utbytet. Främst har lyseringsbufferten mycket stor påverkan på extraktionens utfall men även faktorer som temperatur och inkubationstider är viktiga parametrar som påverkar. Dessutom har lyseringsvolymen en mycket stor inverkan på den slutliga koncentrationen av DNA i extraktet. Om volymen är mindre blir DNA:t mer koncentrerat. Lyseringsbuffertens uppgift är att optimera frisättningen av DNA genom att effektivt lysera cellerna. Detta uppnås genom att kombinera kemiska faktorer som detergenter (t.ex. TWEEN20 som används idag på Nationellt forensiskt centrum (NFC) eller natriumdodecylsulfat (SDS)) och enzymatiska faktorer (t.ex. proteinas K) [9]. TWEEN 20 och SDS verkar genom att göra proteiner och lipider lösliga, vilket leder till att cellmembranet, som till största delen består av dessa komponenter, löses upp [10]. Dessutom denaturerar detergenterna DNA-bindande proteiner som histoner vilket gör att DNA:t enklare kan frisättas. Enligt tidigare studier har användning av SDS visat högre utvinning av DNA [11], vilket beror på att SDS är en starkt laddad detergent [10].
Dessutom, genom att addera fysikaliska faktorer (inkubationer) och mekaniska faktorer (vortex) under extraktionerna har DNA-utbytet kunnat förbättrats ytterligare [12].
DNA-kvantifiering med kvantitativ PCR (qPCR)
Inom forensisk DNA-analys bestäms DNA-kvantitet och kvalitet med qPCR efter slutförd extraktion. DNA-kvantifiering sker dels för att bestämma huruvida provet bör spädas innan amplifiering i multiplex-PCR, för att undvika
DNA-överbelastning om koncentrationen är för hög och dels för att exkludera prover med alltför låg DNA-koncentration. Till skillnad från konventionell PCR där detektion och kvantifiering görs med gelelektrofores efter fullbordad PCR, minimeras hanteringen av post-PCR produkter vid användning av qPCR. Tack vare att qPCR innefattar ett fluorescensdetektionssystem blir analysen snabbare och effektivare. Detta leder också till att qPCR har högre känslighet och
reproducerbarhet [1,13].
qPCR består av temperaturcykler. Första fasen i en temperaturcykel (denatureringsfasen) börjar med att temperaturen höjs (vanligen 95°C) i
instrumentet, vilket får allt dubbelsträngat DNA (dsDNA) att denaturera och bli enkelsträngat (ssDNA). I nästa fas (vanligen kring 60°C, men beroende på smälttemperaturen hos primers) binder korta och specifika DNA-sekvenser (primers) in till ssDNA (annealingfasen) och fungerar som startläge för DNA-polymerasets syntetisering. I sista fasen (extensionsfasen, vanligen 60 – 72°C) kopierar DNA-polymeraset upp nya komplementära DNA-strängar genom att addera deoxynukleosidtrifosfat (dNTP) till primrarna som ovan nämnt fungerar som ”startläge” för syntetiseringen och primrarna blir en del av amplikonet. Dessa 3 temperaturfaser upprepas under PCR-reaktionen i vanligen upp till 40 cykler, då DNA-kvantiteten är tillräcklig [1]. För att bestämma mängden DNA i slutet av
7
varje cykel i qPCR-analysen krävs sekvensspecifika prober som är konjugerade till en fluorofor (t.ex 6-carboxyfluorescein (6-FAM)) i ena änden och till en quencher-molekyl (t.ex Black Hole Quencher (BHQ)) i andra änden. Vid användning av en sådan hydrolysprob hybridiserar proben till en av DNA-strängarna mellan primrarnas inbindningsställen, i annealingfasen. När temperaturen höjs (extensionsfasen) degraderas så småningom proben av polymerasets 5´exonukleasaktivitet. Medan proben är intakt hindras emissionen från fluoroforen via quenchern genom ”fluorescence resonance energy transfer” (FRET). Men när proben klyvs separeras fluoroforen från quenchern vilket resulterar i en amplifieringsberoende ökning i fluorescens [1,14]. Resultatet erhålls i Cq-värden (cycle of quantification) som indikerar när fluorescensen passerat tröskelvärdet för detektion jämfört med bakgrunden. Mängden DNA fördubblas teoretiskt vid varje cykel under den exponentiella fasen. Om skillnaden mellan två prov är 2 cykler har det ena provet 4 gånger mer ursprungligt DNA. Genom att inkludera standarder med kända koncentrationer kan mängden DNA beräknas. Detta sker genom att logaritmera standardkoncentrationerna mot dess Cq-värden. Därefter kan kvantiteten hos proverna beräknas genom 10^((𝐶𝑞 − 𝑚)/𝑘) som erhålls från standardkurvan [3].
PCR-inhibering. Inhibitorer som stör PCR-analysen kan följa med provet från början eller tillsättas i extraktionsprocessen. Då den exakta mekanismen hos inhibitorer ofta är okänd, klassificeras de baserat på vilken del i PCR-analysen de påverkar; DNA-polymerasinhibitorer, nukleinsyrainhibitorer eller
fluorescensinhibitorer (vid användning av qPCR). DNA-polymerasinhibitorer påverkar enzymet antingen direkt (genom att binda till polymeraset) eller indirekt (genom att otillgängliggöra exempelvis Mg2+). Nukleotidinhibitorer fungerar på så sätt att de blockerar amplifiering genom att binda till primrar, ssDNA eller genom att degradera nukleinsyror. Fluorescensinhibitorer påverkar detektionen av
amplikonen genom att blockera fluorescensen (bildande av fällningar), påverka fluoroforen, störa funktionen hos proben eller 5´-exonukleasaktiviteten hos polymeraset. Inhibitorer kan bestå av både oorganiska (t.ex. kalcium) och
organiska ämnen (t. ex urea, SDS, EDTA eller cellulosa men även proteiner som myoglobin, kollagen och immunoglobin G) [9]. För att lösa inhibitionsproblem går det att späda provet och på så vis späda ut PCR-inhibitorerna [15]. Detta leder dock också till lägre koncentrationer av DNA, vilket inte är önskvärt. Istället för att späda provet kan man applicera pre-PCR behandling, ett koncept som
utvecklats vid Teknisk mikrobiologi (TMB) i Lund, och som innebär att både DNA-extraktion och PCR-analys optimeras och anpassas för att kunna hantera provets specifika inhibitorer. Syftet med pre-PCR behandling är att säkerställa optimal och högkvalitativ amplifiering genom att generera homogena DNA-extrakt med hög koncentration av DNA och låg koncentration av PCR-inhibitorer [3]. Dessutom anpassas reagenserna i PCR-analysen till DNA-extraktets innehåll, till exempel genom att välja ett mer hämmartåligt DNA-polymeras eller ändra pH, jon-koncentration och närvaro av PCR-facilitatorer (t.ex bovint serum albumin som binder inhibitorer som bl.a. hemgrupper, fenoler och melanin) i
buffertsystemet. På så vis kan amplifieringseffektiviteten och PCR-inhibitionstoleransen ökas väsentligt.
DNA-profilering
Efter DNA-kvantifiering följer en multiplex-PCR för DNA-profilering med fluorescensinmärkta primers och kapillärelektrofores där DNA-fragmenten kan identifieras efter längd och inmärkt fluorescensfärg [3]. Vid multiplex-PCR
8
amplifieras 15 STR markörer och en könsmarkör samtidigt i en och samma reaktion. STR-markörer utgör en möjlighet till individuell identifiering (DNA-profilering) inom forensik. STR-sekvenser finns utspridda i hela genomet och består av korta sekvenser (3-6 baspar) i olika antal repetitioner hos olika individer. I och med att varje individ har 2 alleler (en för varje förälder) för varje STR-markör, leder de möjliga kombinationerna till att varje fullständig profil är unik. Längden på de 16 markörerna som amplifierats i varje prov visualiseras i ett elektroferogram som utvärderas kvalitativt.
Optimering av DNA-extraktionsmetoder
2018 analyserades ca 60 000 brottsplatsprover på alla NFC:s driftställen över Sverige1. På NFC använder man chelex-baserade extraktionsprotokoll för
majoriteten av provtyper som bomullstops och cigerettfilterpapper m.m. Vid den forensiska gruppen på TMB i Lund bedrivs ett ständigt metodutvecklingsarbete i nära samarbete med NFC. Via tidigare arbete på TMB har det chelex-baserade extraktionsprotokollet utvecklats och optimerats för att i framtiden möjliggöra en mer effektiv analys och ett ökat antal genererade DNA-profiler. Tidigare studier har visat att DNA-extraktioner av DNA-material från bl.a. tops och
cigarettfilterpapper går att effektivisera och förenkla med tillsatser av detergenter, lyseringsvolymer och inkubationssteg (tid, skak, temperatur osv.). Det har även observerats att val av extraktionsrör har betydelse då det bl.a. tillåter en
reducering av volym vilket ger högre DNA-koncentration [11,16–17]. Exempelvis har det visats att autolys A-rör med SDS som detergent ger högre DNA utbyte vid extraktioner av cigarettfilterpapper och kräver mindre manuellt arbete än dagens metod [11,17]. Autolys A-rör är ett rör-i-rörsystem som tillåter ett smidigt avlägsnande av större material i ett slutet system [18]. Dessutom ges möjligheten att minska volymen från ca 600 µl, som används idag med mikrofugfrör, till ca 200 µl [11,17]. Dock är autolys A-rör inte anpassade för manuell rutinanalys, då de innehar ett skruvlock, vilket gör dem mindre attraktiva för NFC, där analyserna idag utförs manuellt2.
Syfte
Syftet med studien var att utifrån tidigare resultat kring spårsäkringsteknik och extraktionsprotokoll, kombinera ihop dessa till ett slutligt protokoll som är optimalt för de vanligast förekommande brottsplatsproverna.
Etisk bedömning
Ingen etisk bedömning var relevant i denna studie då endast saliv har använts som DNA-källa. Saliven donerades av en frivillig person och provet insamlades icke-invasivt och endast en gång under arbetets gång. I arbetet utfördes heller inga diagnostiska analyser och därmed erhölls ingen känslig information.
1 Personlig kommunikation J.Hedman 2 Personlig kommunikation J.Hedman
9
METOD OCH MATERIAL
Saliv som DNA-källa
Saliv donerades från endast en person vars DNA-profil tidigare var inlagd i NFC:s eliminationsdatabas, genom att denne fick spotta i ett falconrör. Därefter späddes saliven med 0,9 % NaCl till 6 olika spädningar (1:2, 1:5, 1:10, 1:20, 1:40, 1:80). Respektive spädning alikvoterades ut i 15 st 1,5 ml mikrofugrör med 0,5 ml saliv i varje rör. Dessa 90 rör med saliv fungerade som en ”salivbank” för alla tester som utfördes inom detta arbete.
Beredning och säkring av forensiska spår
Inför alla försök utfördes en spårberedning. 20 µl saliv av vald
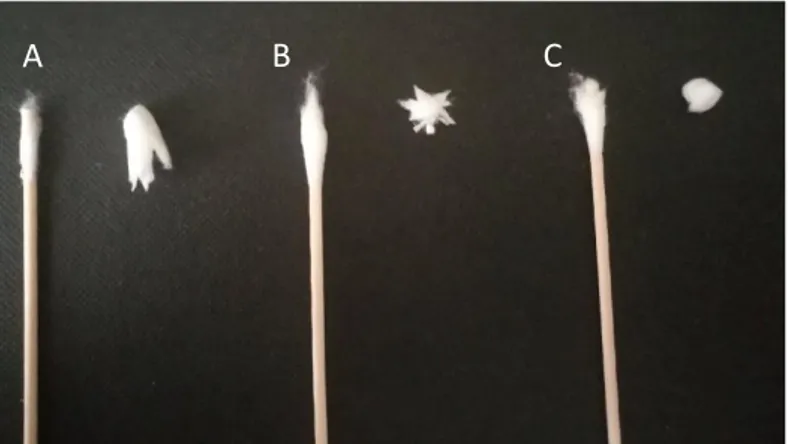
spädning/spädningar applicerades på en glasyta som mätte 2x2 cm. Exempelvis, 20 µl av 1:20-batchen innehåller 1µl saliv. Saliven tilläts därefter torka över natten. Innan spårsäkring av saliven från glasytan utfördes pipetterades 40 µl 0,9% NaCl, jämnt distribuerat till övre delen av bomullstopsen (Cotton Tipped Applicator, SELEFA, Helsingfors, Finland), som är den aktuella topsen vid spårsäkring av brottsplatser. Vid spårsäkringen var målet att absorbera saliven homogent på topsens övre halva genom att rotera topsen, i en ca 60 graders vinkel, samtidigt som topsen fördes i en svepande rörelse som garanterar att hela fläcken topsas. Efter spårsäkring tilläts topsen torka en timme innan bomullen klipptes ner i extraktionsrör. För klippning av bomullstopsen fattades saxen i en upprätt position och bomullen klipptes sedan loss från träpinnen genom att klippa sidorna nerifrån mot toppen (likt en blomma) innan topsen drogs av genom att rotera pinnen (Figur 2a). I vissa fall klipptes övre halvan av topsen rakt av (ÖH) och i andra fall klipptes den yttre delen av den övre halvan (YÖH) av (Figur 2b, c). Rören förvarades i frys (-20°C) i väntan på kvantifiering.
Figur 2. Bomullstopsen, Cotton Tipped Applicator, SELEFA, klippt på tre olika sätt.
Standardklippning enligt metod (A), övre halvan (YÖH) (B) och yttre övre halvan (YÖH) (C).
DNA-extraktion
4 olika protokoll för extrahering ingick i arbetet varav ett var standardprotokoll och används på NFC idag. De resterande tre är protokoll som är framtagna på TMB i Lund. De största skillnaderna mellan metoderna var val av detergent och rör.
ChelexDirekt (NFC) extraktion
Standardextraktionsprotokollet som används på NFC idag utfördes genom att först, efter att bomullen klipptes ner i mikrofugrör, tillsätta 600 µl 5% chelex
B
10
(BioRad Laboratotories, CA, USA) med 0,2% TWEEN 20 (Sigma-Aldrich, St. Louise, USA) och 6 µl proteinas K (10 mg/ml) (Sigma, St. Louise, USA). Efter att alla prover vortexats ca. 5 sek/rör tilläts de inkubera i rumstemperatur i 30 min där rören vortexades (5 sek/rör) efter 15 min och i slutet. Därefter inkuberades
proverna i värmeblock, 56°C i en timme. Efter att proven vortexades ännu en gång (5 sek/rör) inkuberades de 20 min i värmeskåp vid 100°C. När rören svalnat centrifugerades proverna i 11 000g i 1 minut. Slutligen lades proverna i frys (-20°C) för förvaring.
Extraktion med thermoskak
Ordinarie protokoll, d.v.s. ChelexDirekt (NFC) extraktion följdes med undantaget att inkubering i rumstemperatur uteslöts och inkubation i värmeblock ersattes av inkubation i thermoskak (BioShake iQ, Jena, Tyskland) vid 1500 rpm i 60 minuter.
Autolysextraktion med TWEEN 20/SDS
Skillnaden mellan autolysextraktion med TWEEN 20 respektive SDS var just val av detergent. För dessa protokoll tillsattes endast 200 µl 5% chelex med 0,2% TWEEN 20 eller 0,025% SDS och 2 µl proteinas K (10 mg/ml) till autolys A-rör (Hamilton Robotics, Malans, Schweiz). Proven vortexades (5 sek/rör) och läts inkubera i rumstemperatur i 30 min. Efter inkubationen vortexades rören (5 sek/rör) och lades i vattenbad för inkubation i 30 min, 56°C (alternativt i
värmeskåp i ca. en timme i 56°C). Rören togs sedan ut, vortexades (5 sek/rör), och lades in i värmeskåpet igen för ytterligare en inkubation på 100°C i 20 minuter. Efter att rören svalnat lyftes inner-röret i autolys A-röret upp och låstes i en position som tillät filtrering till det yttre röret vid centrifugering (Figur 4). Centrifugeringen utfördes vid 2000g i 10 min. Efteråt slängdes inner-röret
(innehållande bomullen och chelexkulorna) och lysatet i det yttre röret överfördes till ett mikrofugrör för förvaring i -20°C i frys.
Optimering av protokoll
För att optimera det protokoll som ansågs ha störst potential till förbättrat DNA-utbyte, utfördes tester enligt dessa protokoll med olika kombinationer av tops- och rörtyp samt olika klippta bitar av topshuvudet.
Tops
Topsen som testades utöver Selefatops var av olika material och hade därmed olika egenskaper. Topsen bestod av bomullstops (Puritan® Sterile Cotton Tipped Applicators, Puritan Medical Products, ME, USA), flockad tops
(4N6FLOQSwabs® crime scene, Copan Flock Technologies, Brescia, Italy) och skumgummitops (Micro Foam Head, VWR Critical Swab, VWR, West Chester, USA). För bomullstopsen från Puritan och flockad tops var förberedning av spårsäkringen mycket lik förberedning för standardtopsen från Selefa. 40 µl 0,9% NaCl applicerades, jämnt distribuerat, på absorptionsytans övre halva.
Skumgummitopsen doppades till hälften i 0,9% NaCl och drogs sedan av längs kanten av NaCl-röret för att bli av med överskott av vätska. Spårsäkringen utfördes på samma sätt som för Selefa-topsen. Efter spårsäkringen klipptes bomullstopsen från Puritan rakt av genom skaftet strax under bomullen
(absorptionsytan). Optimeringsförsök där puritantopsen klipptes på samma sätt som standardtopsen (Figur 2a) utfördes också. Flockad tops och skumgummitops
11
var konstruerade till att kunna klippas eller brytas av rakt ned i extraktionsrören (Figur 3).
Figur 3. A) från vänster till höger: Bomullstops (SELEFA och Puritan®), flockad tops (4N6 FLOQSwabs®)
och skumgummitops (Micro Foam Head). B). De olika topsen klippta på respektive sätt.
Extraktionsrör
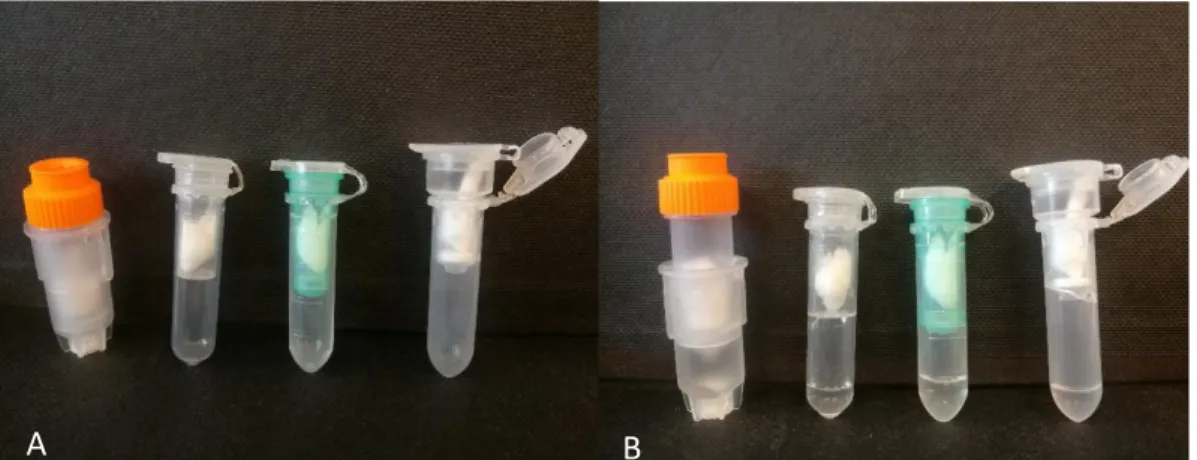
Förutom mikrofugrör och autolys-A-rör utfördes tester med PrepFiler LySep™ Columns (LySep-rör) (Thermo Scientific, Warrington UK), Investigator® Lyse&Spin Baskets (Qiagenrör) (Qiagen, Hilden, Tyskland) och Sampletype i-sep® SQ (iSep-rör) (Biotype Diagnostic GmbH, Dresden, Tyskland) (Figur 4). Dessa rör är alla rör-i-rörsystem men passar mer för manuell hantering till skillnad från autolys A-rören. Extraktionsprotokollet för respektive rör skiljde sig åt vid centrifugeringssteget. För LySep-rören användes en centrifugeringshastighet på 11 000g i 1 minut, medan Qiagenröret centrifugerades vid 20 000g i 1 minut och iSep-röret centrifugerades på 13 400g i 1 minut. Alla extraktioner med dessa rör avslutades med att slänga den inre filterkorgen. Då komplikationer med dessa rör uppstod, såsom att lysatet inte kunde rinna genom den inre korgdelen under centrifugeringen, följde optimeringsförsök för att lösa eller kringgå problemen.
Figur 4: A) Från vänster till höger: Autolys A rör, Qiagenrör, iSep-rör och LySep-rör innan centrifugering. B)
Efter centrifugering.
Undersökning av PCR-hämning
Då ett flertal prover som extraherats i Qiagenröret uppvisade PCR-hämning vid upprepade försök, blev det aktuellt att undersöka vilken eller vilka faktorer som orsakade hämning. Hämmade prover kvantifierades spädda 1:5 med samma
A A B
12
polymeras som de kvantifierades med första gången. Efter det kvantifierades de med ett annat mer hämmartåligt DNA-polymeraskit (PefeCTa qPCR Toughmix, QuantaBio, Beverly, USA).
DNA-kvantifiering med qPCR
Kvantifiering gjordes genom qPCR av retinoblastoma 1 genen (RB1) genom att blanda följande reagens: SuperQ-vatten (Merck millipore, Bedford, USA), 1x Immobuffert (Bioline Reagents, London, UK), 0,2 mM deoxynukleotidtrifosfat (dNTP) (Roche Diagnostics, Basel, Schweiz), 4 mM MgCl2 (Roche Diagnostics) ,
0,3 µM RB1_80 forward primer och RB1_235 reverse primer (Life Technologies, New York, USA), 0,2 µM RB1 prob 6-carboxyfluorescein- (6FAM) (Life
Technologies), 0,1 mg/ml BSA (Roche Diagnostics) och 0,05 U/µL Immolase polymeras (Bioline Reagents). När PCR-protokollet för Toughmix användes, tillsattes endast SuperQ-vatten, 1x Toughmix (Quantabio, Beverly, USA), 0,3 µM RB1_80F primer och RB1_235R-primer och 0,2 µM RB1 prob FAM.
DNA-extrakten tinades varpå 5µl extrakt sattes till 15µl mix i varje PCR-brunn. Brunnarna, med en total reaktionsvolym på 20 µl, analyserades i Light Cycler Nano real-time PCR instrument med Light Cycler Nano Software 1.1(Roche Diagnostics). För att kunna utföra DNA-koncentrationsbestämning i proverna inkluderades, i duplikat och ibland triplikat, Quantifier Human DNA Standard (ThermoFisher Scientific, Waltham, USA) i en spädningsserie av 1:10 (10 - 0,01 ng/µl) där 5 µl av respektive spädning sattes till 15 µl mix. I PCR-programmet för Immolase och RB1-analys inkluderades ett initialt aktiveringssteg vid 95°C i 10 minuter följt utav 45 cykler innehållande tre faser: denaturering vid 95°C i 10 sek, primer-annealing vid 60°C i 20 sek och extension vid 72°C i 30 sek. I PCR-programmet för Thoughmix-analys initierades programmet med ett aktiveringssteg på 95°C i 3 minuter följt utav 45 cykler innehållande två faser: denaturering vid 95°C i 10 sek och primer annealing/extension vid 60°C i 50 sek. Försök på NFC
Vid ett tillfälle utfördes ett större försök på NFC i Linköping med samma spårsäkring och extraktions-protokoll som beskrivet ovan men med NFC:s validerade analysmetoder. Istället för kvantifiering med det ovan beskrivna PCR-protokollet användes QuantifilerTM Hp DNA Quantification Kit (ThermoFisher
Scientific, Waltham, USA) med ABI 7500 Real-time PCR system (ThermoFisher Scientific) istället för Light Cycler Nano real- time PCR instrument. Efter
kvantifieringen utfördes en multiplex PCR med 16 fluorocensinmärkta markörer (enligt NFC rutin) i GeneAmp®PCR System 9700 (Thermo Fisher Scientific). Till 7,5 µl PCR-mix tillsattes 17,5 µl extrakt. DNA-fragmenten (markörerna) separerades med kapillärelektrofores på Applied Biosystems 3500xl Genetic Analyzer (Thermo Fisher Scientific) varpå ett elektroferogram erhölls för analys. Resultatet med avseende på den totala topphöjden jämförelse med
kvantifieringsresultaten. Iscensatt brottsplats
En iscensatt brottsplats upptäcktes med 6 olika typer av spår bestående av fem fingeravtryck på ett fönster (tre av dessa fingeravtryck med blod), en läskburk, en penna, en telefon, två hylsor och en kopp. Tre fingeravtryck säkrades med
Puritantops och två med Selefatops enligt beskrivning i ”Beredning och säkring av forensiska spår”. Koppen säkrades på kanten och örat med två olika
13
Puritantops. Telefonen säkrades med två Puritantops; en för knapparna och en för telefonens övriga yta. Pennan spårsäkrades genom att använda Puritantops; en för ”handtaget” och en för toppen av pennan ”klicken”. Läskburken säkrades med Puritantops på drickytan. De 2 hylsorna säkrades med 4N6FLOQ enligt framtaget protokoll respektive Puritantops. Topsen tilläts torka en timme i rumstemperatur. För Puritantops och Selefatops klipptes övre halvan av bomullen medan den flockade topsen (4N6FLOQ) bröts av enligt metod. Observera att för
fingeravtryck med synlig blodfläck klipptes endast en liten bit av topsen (mindre än övre halvan) medan resten av topsen extraherades i ett eget rör. SDS-
extraktion med Qiagenrör användes vid detta utförande vilket kräver en lyseringsvolym på endast 200 µl. Kvantifieringen utfördes enligt beskrivning i ”DNA-kvantifiering med qPCR”.
Analys av resultat
Alla resultat lades in i Excel där medelvärde och standardavvikelse räknades ut och visualiserades i stapeldiagram med standardavvikelse. För att visualisera skillnaderna mellan de olika grupperna i NFC-försöket utformades ett låddiagram där medianvärde, övre och undre kvartilen, samt minimum- och maximumvärde visas för respektive försöksgrupp.
RESULTAT
Inledningsvis undersöktes det vilka protokoll som har störst potential att förbättra och förenkla dagens extraktionsprotokoll på NFC. Dessa protokoll utvärderades och jämfördes därpå mot dagens protokoll på NFC (referensprotokoll). Då spårsäkringen utförts på samma sätt för samtliga protokoll antas mängden DNA som spårsäkrats för varje protokoll inom varje spädning vara mycket lika. Val av protokoll
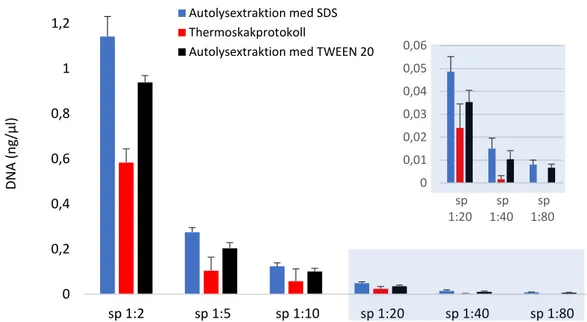
Bland de tre framtagna protokollen; autolysextraktion med SDS, autolysextraktion med TWEEN 20 och thermoskak-protokoll, var extraktion med SDS det
effektivaste protokollet med avseende på DNA-koncentration efter extraktion (Figur 5). Vid extraktion av 10 µl saliv (spädning 1:2) resulterade autolys-extraktion med SDS i en DNA-koncentration på 1,142 ± 0,089 ng/µl (n = 3) medan thermoskak-protokollet och autolysextraktion med TWEEN 20 gav 0,583± 0,061 ng/µl respektive 0,939± 0,029 ng/µl. Detta gällde också för tester med lägre mängder saliv. För autolysextraktion med SDS erhölls, vid extraktion av 0,5µl saliv (spädning 1:40), en DNA- koncentration på 0,015± 0,004 ng/µl, jämfört med thermoskak-protokollet och autolysextraktion med TWEEN 20 som gav 0,002± 0,002 ng/µl respektive 0,010± 0,004 ng/µl.
14 Figur 5. Medelvärde och standardavvikelse för DNA-koncentrationen av tre protokoll som är framtagna på
TMB i Lund, undersökt på sex olika salivspädningar (sp) med fallande koncentration, (n = 3).
Val av extraktionsrör
I Figur 6 kan två alternativa rör till autolys A rör betraktas; Qiagenrör och LySep-rör. Alla prover är extraherade med SDS-protokollet. Vid spårsäkring av 10 µl saliv (sp 1:2) gav extraktion med Qiagenröret en DNA-koncentration på 1,100 ± 0,065 ng/µl (n=3). Extraktion av samma mängd saliv med autolysröret,
kvantifierades till 1,188 ± 0,254 ng/µl (n=3). Dock resulterade proven som extraherats i Qiagenrör i ett hämmat prov vid spädning 1:2 och flera vid spädning 1:10. Betydligt fler prov som extraherats med LySep-rören var hämmade.
Autolysrören (Figur 6) gav kvantifieringsresultat som var lika tidigare resultat (Figur 5) för de båda salivkoncentrationerna som testades. Inga fler försök
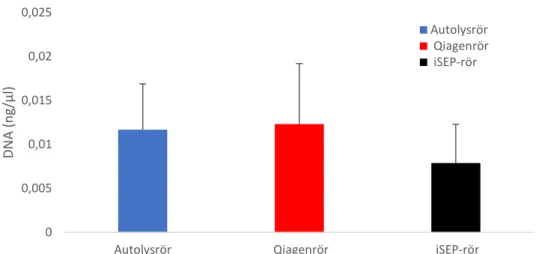
utfördes med LySep-rören efter detta. Däremot avser Figur 7 ett test med ett tredje alternativ; iSep-rör. Dessa prover kvantifierades utan PCR-hämning. Spårsäkring av 1 µl saliv erhöll en DNA-koncentration, efter extraktion med iSep-rör, på 0,008± 0,004 ng/µl.
Figur 6. Medelvärde och standardavvikelse för DNA-koncentrationer från tre olika rör utförda med
SDS-extraktion på två olika spädningar (sp, n = 3). OBS: n = 2 för Qiagen i spädning 1:10 och n = 0 för Lysep i spädning 1:10 pga PCR-hämning.
0 0,2 0,4 0,6 0,8 1 1,2 sp 1:2 sp 1:5 sp 1:10 sp 1:20 sp 1:40 sp 1:80 DN A (n g/µl) Autolysextraktion med SDS Thermoskakprotokoll
Autolysextraktion med TWEEN 20
0 0,2 0,4 0,6 0,8 1 1,2 1,4 1,6 sp 1:2 sp 1:10 DN A (n g/µl) Autolysrör Qiagenrör Lyseprör 0 0,01 0,02 0,03 0,04 0,05 0,06 sp 1:20 sp 1:40 sp 1:80
15
Figur 7: Medelvärde och standardavvikelse för DNA-koncentration från tre olika rör utförda med SDS-extraktion på 1 µl saliv (1:20). Försöket utfördes i 4 replikat (n=4) för respektive rör. På grund av hämning är endast n = 2 för Qiagenrören.
Undersökning av PCR-hämning
Vid ett test av Qiagenrör med SDS- respektive NFC-protokoll var resultatet ojämförbart då majoriteten av extrakten inom NFC-protokollet var hämmade vid kvantifieringen (data visas ej). Då tidigare hämmade extrakt kvantifierades med toughmix, återgavs DNA-koncentrationer för dessa extrakt utan hämning. I tabell 1 kan dessutom de hämmade extrakten jämföras ospädda och spädda, med
standard qPCR-reaktion (Immolase DNA polymeras). De ospädda proverna resulterade än en gång i blanka resultat och de spädda kunde kvantifieras. Från dessa två tester är det tydligt att det som stör kvantifieringen dämpas när extrakten späds (tabell 1).
Tabell 1: Tidigare hämmade kvantifieringar vid extraktion med Qiagenrör (prov 2-7). Alla prover
är kvantifierade i samma qPCR reaktion (qPCR-protokoll för RB_1 analys med Immolase), ospädda och spädda (1:5) med Super-Q-vatten. Resultaten är angivna som Cq-värden. För prov 1-3 var 10 µl saliv spårsäkrat. För prov 4-7 var en salivmängd på 2 µl spårsäkrad.
*1 2 3 4 5 6 7
Ospädd 28,175626 - - - -
Spädd 30,451620 32,841457 32,92862 34,19085 34,97779 35,716996 34,77484
* Kontroll, detta prov har inte tidigare erhållit hämmat resultat.
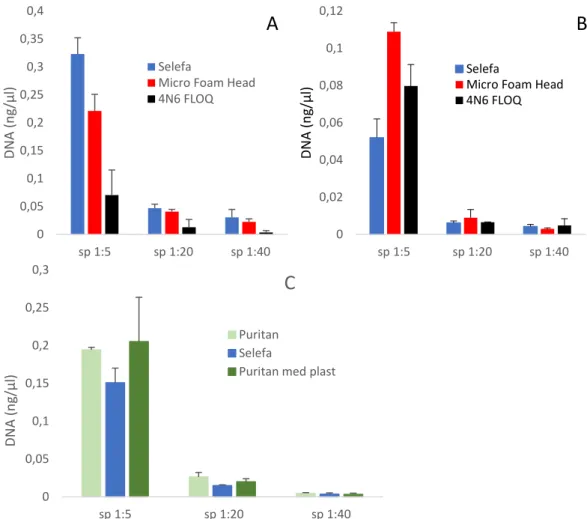
Val av tops
Vid analys av alternativ till Selefatops indikerar resultaten (Figur 8a) att Selefatops vid extraktion i autolysrör med SDS är att föredra då det ger högst DNA-koncentration för alla testade mängder av saliv. Vid jämförelse mellan SDS-extraktion (Figur 8a) och NFC-protokollet (Figur 8b) är det tydligt att
resultaten skiljer sig väsentligt åt för Selefatops, men inte för övriga 2 tops (Micro Foam Head respektive 4N6 FLOQSwab). Överlag resulterade SDS-protokollet dock i högre koncentrationer DNA jämfört med NFC-protokollet (Figur 8a - b). Figur 8c visar resultat från ett test med ytterligare ett alternativ till Selefatops i form av en annan bomullstops, med plastskaft (Puritan® Sterile Cotton Tipped Applicators). Vid användning av Selefatops för 4µl saliv erhölls en DNA-koncentration på 0,151±0,019 ng/µl, men denna DNA-koncentration kunde ökas till 0,194±0,003 ng/µl när spårsäkringen skedde med Puritantops och bomullen klipptes av från plastskaftet (likt Selefa (Figur 3a)). Då hela bomullshuvudet,
0 0,005 0,01 0,015 0,02 0,025
Autolysrör Qiagenrör iSEP-rör
DN A (n g/µl) Autolysrör Qiagenrör iSEP-rör
16
inklusive plastskaftet som bomullen är lindad runt, klipptes ner i extraktionsröret, ökade DNA-koncentrationen till 0,205±0,059 ng/µl. Alla extraktioner i detta försök utfördes i triplikat.
Figur 8. Medelvärde och standardavvikelse för DNA koncentration från tre olika tops, extraherade för tre
olika salivspädningar (sp). DNA är extraherat enligt följande metoder A) SDS-protokoll i autolys A-rör. B) NFC protokoll i mikrofugrör. C) SDS-protokoll i autolys A-rör där puritantopsen är klippt på två vis enligt metodbeskrivning, bara bomullen (Puritan) och hela bomullen med plastpinne (Puritan med plast) (Figur 3b).
Kombinationstester
I nästa försök kombinerades tre olika rör (mikrofugrör, autolys-A-rör och Qiagenrör) med två olika topsar (Selefatops, Puritantops klippt rakt av) och två olika extraktionsprotokoll (NFC-, SDS-protokoll) för extraktion av 1µl saliv (Figur 9). Mikrofugrör inkluderades endast för NFC-protokollet, där volymen lyseringsbuffert är 600 µL jämfört med 200 µL som används vid extraktion med Autolysrör och Qiagenrör. Resultaten visar precis som i föregående försök att SDS-protokollet har en positiv effekt på DNA-utbytet från Selefatops. Denna effekt av SDS verkar dock försvinna då Puritantops används. Vid jämförelse mellan Selefatops och Puritantops med SDS och autolysrör erhölls högre DNA-mängd från Puritantops (0,043± 0,019 ng/µL) jämfört med Selefatops (0,023± 0,001 ng/µL). Vid användning av autolysrör med NFC-extraktion kunde DNA-koncentrationen ökas från 0,0128±0,0013 ng/µl med Selefatops, till 0,046±0,008 ng/µl med Puritantops. Extraktion i Qiagenrör gav generellt höga
DNA-koncentrationer, men det bör observeras att flera av dessa prover orsakade PCR-hämning. 0 0,05 0,1 0,15 0,2 0,25 0,3 0,35 0,4 sp 1:5 sp 1:20 sp 1:40 DN A (n g/µl) Selefa
Micro Foam Head 4N6 FLOQ 0 0,02 0,04 0,06 0,08 0,1 0,12 sp 1:5 sp 1:20 sp 1:40 DN A (n g/µl) Selefa
Micro Foam Head 4N6 FLOQ 0 0,05 0,1 0,15 0,2 0,25 0,3 sp 1:5 sp 1:20 sp 1:40 DN A (n g/µl)
C
Puritan SelefaPuritan med plast
17 Figur 9. Medelvärde och standardavvikelse (n=3) för tre olika rör, två olika topsar och två olika
extraktionsprotokoll för 1µl saliv. Puritantopsen är klippt från skaftet och Selefatopsen enligt NFC-metod.
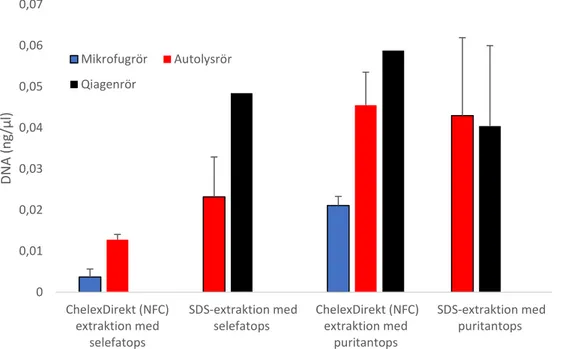
Försök på NFC, Linköping
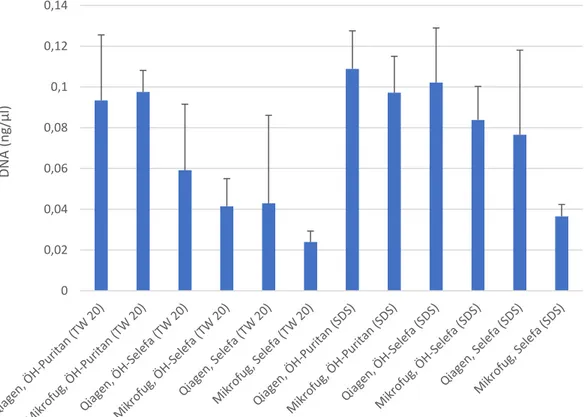
Ett försök där olika extraktionsprotokoll jämfördes, utfördes på NFC i Linköping (Figur 10). Metoderna utfördes enligt beskrivning, och kvantifieringen gjordes med NFC:s rutinprotokoll för qPCR-analys. Fyra olika rör (mikrofugrör,
Qiagenrör, iSep-rör och autolys-A-rör), två olika tops (Selefatops och Puritantops, varav Selefatops är klippt på tre olika sätt) samt två olika lyseringsbuffertar (NFC och SDS) kombinerades på olika sätt (n=5), för en salivmängd på 2 µl. Två tidigare otestade grupper infördes i detta försök; Selefatops klippt som ”övre halva” (ÖH) respektive ”yttre övre halva” (YÖH) extraherad med NFC-protokoll i mikrofugrör. Eftersom mindre mängd bomull klipptes ned i dessa prover, kunde mindre mängd lyseringsbuffert tillsättas, vilket ökar DNA-koncentrationen. Högst DNA-koncentration erhölls vid spårsäkring med Puritantops som extraherats med NFC-protokoll i Qiagenrör (0,047±0,015 ng/µl). Lägst DNA-koncentration erhölls för NFC:s referensmetod (Selefatops, extraherat med NFC-protokoll i
mikrofugrör) med 0,003± 0,001 ng/µl. Överlag gav Selefatops (klippt på normalt vis) tillsammans med NFC-protokoll lägst DNA-utbyte oavsett rör, medan Puritantops erhöll de högsta koncentrationerna för majoriteten av rören. Endast extraktion av den yttre övre halvan av Selefatopsen i mikrofugrör gav högre utbyte jämfört med Puritantops extraherad med samma protokoll, troligtvis till följd av den reducerade volymen som sattes till den yttre övre halvan Selefatops. Då Selefatopsen extraherades med SDS-protokoll erhölls också betydligt högre DNA-mängder jämfört med NFC-protokollet, oavsett rör.
Det observerades PCR-hämning för flera prover med Selefatops som extraherats med NFC-protokoll, oavsett rör. Flest hämmade prover observerades för
Selefatops i autolys A-rör med NFC-protokoll (fyra av fem replikat). För Selefatops i iSep-rör med NFC-protokoll var två av fem prover hämmade. För Selefatops i mikrofugrör respektive Qiagenrör med NFC-extraktion var ett prov från vardera grupp på fem prover hämmat.
0 0,01 0,02 0,03 0,04 0,05 0,06 0,07 ChelexDirekt (NFC) extraktion med selefatops SDS-extraktion med selefatops ChelexDirekt (NFC) extraktion med puritantops SDS-extraktion med puritantops DN A (n g/µl) Mikrofugrör Autolysrör Qiagenrör
18 Figur 10. Jämförelse av DNA-utbyte för olika extraktionskombinationer utförda i 5 replikat. 1µl saliv
spårsäkrades. De testade komponenterna anges i följande ordning: rör, tops, protokoll, volym. Ingen tops= Selefatops, Pur= Puritantops, ChD= ChelexDirekt
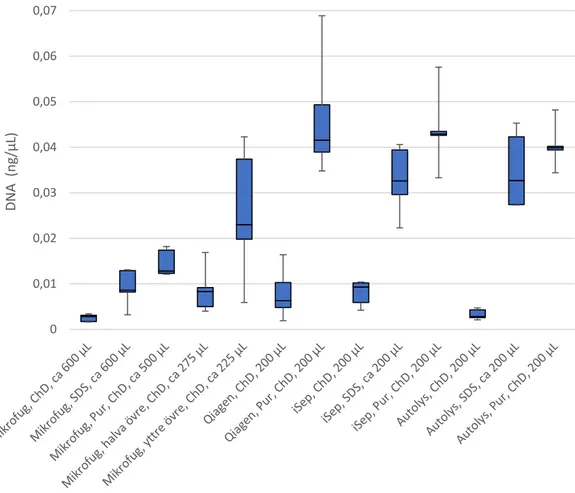
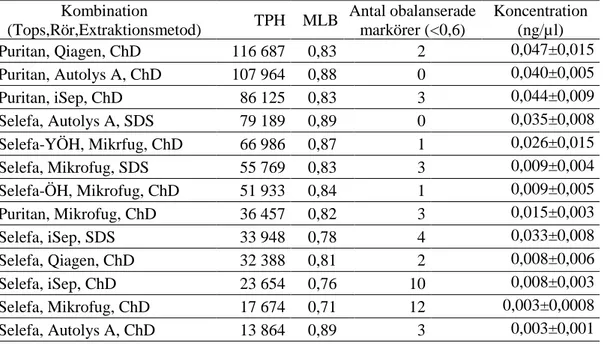
Från NFC-försöket valdes det prov med näst och tredje högsta DNA-koncentration ut från varje grupp till STR-typning (Tabell 2). Högst
medeltopphöjd registrerades för NFC-protokoll med Puritantops i Qiagenrör. Puritantops resulterade i de tre högsta medeltopphöjderna medan Selefatops gav de fem lägsta medel-topphöjderna. För de hetrozygota markörerna var den lokala balansen (MLB) mellan de respektive markörerna jämnast för SDS-extraktion med Selefatops i autolys A-rör (total balans = 1). Överlag följer höga topphöjder de höga koncentrationerna. För de kombinationer som gav minst antal
obalanserade markörer var autolys A-rör den enda gemensamma faktorn.
0 0,01 0,02 0,03 0,04 0,05 0,06 0,07 DN A (n g/µL )
19
Tabell 2: Extraktionskombinationer som visas i topphöjdsordning (högst till lägst) med
information om MLB, antal obalanserade markörer och koncentration efter kvantifiering med qPCR. Kombination (Tops,Rör,Extraktionsmetod) TPH MLB Antal obalanserade markörer (<0,6) Koncentration (ng/µl) Puritan, Qiagen, ChD 116 687 0,83 2 0,047±0,015 Puritan, Autolys A, ChD 107 964 0,88 0 0,040±0,005 Puritan, iSep, ChD 86 125 0,83 3 0,044±0,009 Selefa, Autolys A, SDS 79 189 0,89 0 0,035±0,008 Selefa-YÖH, Mikrfug, ChD 66 986 0,87 1 0,026±0,015 Selefa, Mikrofug, SDS 55 769 0,83 3 0,009±0,004 Selefa-ÖH, Mikrofug, ChD 51 933 0,84 1 0,009±0,005 Puritan, Mikrofug, ChD 36 457 0,82 3 0,015±0,003 Selefa, iSep, SDS 33 948 0,78 4 0,033±0,008 Selefa, Qiagen, ChD 32 388 0,81 2 0,008±0,006 Selefa, iSep, ChD 23 654 0,76 10 0,008±0,003 Selefa, Mikrofug, ChD 17 674 0,71 12 0,003±0,0008 Selefa, Autolys A, ChD 13 864 0,89 3 0,003±0,001 ChD= ChelexDirekt (NFC) protokoll YÖH= Yttre-övre halvan av topsen är klippt ÖH= Övre halvan av topsen är klippt TPH= Topphöjd
MLB= Mean local balance
Utvärdering av klippning för Puritantops
Vid extraktion av Puritantopsen (hela topsen, 2/3 av topsen och 1/3 av topsen), för att undersöka möjligheten att eventuellt klippa övre halvan likt Selefatopsen, finns resultatet presenterat i Figur 11 med både NFC och SDS-protokoll. Alla
extraktioner utfördes i mikrofugrör. Här kunde ingen positiv effekt av SDS observeras jämfört med NFC-protokollet, och vid samma volym (200 µL) erhölls likvärdiga koncentrationer mellan Selefatops och Puritantops.
DNA-koncentrationerna från Puritantopsen ökade som förväntat ju mindre bomull, och därmed lyseringsvolym, som användes.
Figur 11: Puritantops klippt på 3 olika sätt (hela topsen, 2/3 av topsen och 1/3 av topsen), extraherad med NFC-protokoll respektive SDS-protokoll. Selefatops där övre halvan är klippt finns med som referens då detta testats tidigare. Extraktionen utfördes på 2µl saliv.
0 0,02 0,04 0,06 0,08 0,1 0,12 Puritan avklippt -600µl Puritan 2/3-300µl Puritan 1/3-200µl Selefa ÖH-200µl DN A (n g/µl) ChelexDirekt SDS-protokoll
20 Slutlig jämförelse av de mest lovande extraktionsprotokollen
I ett sista försök jämfördes följande extraktionsparametrar: Qiagenrör mot mikrofugrör, Selefatops mot Puritantops då båda tops klippts som ”övre halva” och NFC- mot SDS-protokoll (n = 4) (Figur 12). Selefatops, klippt på dagens vis, inkluderades som referensmetod, där 600 µL chelex-lösning krävdes för att täcka bomullen. Alla övriga prover extraherades med 200 µL chelex-lösning. Överlag genererar extraktion av både Puritantopsens och Selefatopsens övre halva ett högre DNA-utbyte än när Selefatopsen klipps enligt dagens metod. När Selefatopsens övre halva (ÖH) extraherats med NFC-protokoll i Qiagenrör kvantifierades DNA-koncentrationen till 0,059±0,032 ng/µl. När motsvarande kombination av tops och rör extraherades med SDS-protokoll, kvantifierades DNA-koncentrationen till 0,100±0,027 ng/µl. Vid användning av Puritantops var dock resultaten mellan SDS- och NFC-protokoll likvärdiga. Extraktion med Qiagenrör gav generellt något högre DNA-utbyte jämfört med extraktion i
mikrofugrör då samma volym chelexlösning användes, men skillnaderna var små. För både NFC- och SDS-protokollet gav 2 prover dessutom PCR-hämning då Selefatops extraherades med Qiagenrör.
Figur 12. Olika kombinationer av rör, tops, klippning och extraktionsprotokoll. För Puritan är endast
övre-halvan (ÖH) klippt och för selefa redovisas klippning av övre-övre-halvan och standardklippning som referens. 2 µl saliv spårsäkrades för dessa extraktioner.
Iscensatt brottsplats
Efter att ha utvärderat vilken eller vilka protokoll som är att föredra iscensattes en brottsplats för att bestämma om valda protokoll är kompatibla med spår på vanligt förekommande föremål på brottsplatser (tabell 4). För fingeravtrycken
kvantifierades alla prover, med och utan blod. För tre avtryck, innehållande blod, extraherades även resten av topsen vilket resulterade i ett PCR-hämmat prov. Bland alla prover gav endast ett prov ett blankt resultat; extraktion av toppen av pennan, ”klicken”. Liknande koncentration erhölls vid spårsäkring av hylsa med 4N6FLOQ-topsen och Puritantopsen.
Tabell 4: Kvantifiering av spår hittat på en iscensatt brottsplats.
0 0,02 0,04 0,06 0,08 0,1 0,12 0,14 DN A (n g/µl)
21
Prov.nr Typ av spår, Tops ng/µl DNA
1 Fingeravtryck (blod), Puritan resten av 1:an
0,030
2 0,034
3 Fingeravtryck, Puritan 0,014
4 Fingeravtryck (blod), Selefa Resten av 4:an
0,024
5 0,027
6 Fingeravtryck (blod), Selefa Resten av 6:an
0,013
7 0,001
8 Fingeravtryck, Puritan 0,016
9 Läskburk, Puritan 0,521
10 Tele (knappar), Puritan 0,256
11 Tele, Puritan 0,228
12 Penna (toppen), Puritan -
13 Penna handtaget, Puritan 0,027
14 Kopp, Puritan 0,157
15 Kopp (handtag), Puritan 1,208
16 Hylsa, Puritan 0,153
17 Hylsa, 4N6FLOQ 0,153
DISKUSSION
För att säkra DNA-spår från ytor och hårda föremål används idag Selefatops på NFC och av polisen runt om i Sverige. Oavsett var spåret säkras skickas topsen till NFC för klippning och DNA-extraktion. Då personen som klipper topsen inte alltid vet hur spårsäkringen är utförd, d.v.s. om spåret sitter på toppen av topsen eller runtom hela bomullshuvudet, klipps idag hela det yttre lagret av bomullen av från topsen för att inte riskera att exkludera befintliga DNA-spår. Då klippningen är något krånglig och utförs av olika personer, resulterar detta i stor variation i hur stor och fluffig den avskalade bomullen blir. Dessutom kan selefatopsen variera i storlek från början. Allt detta leder till att lyseringsvolymen måste anpassas enskilt för varje prov för att provet ska täckas helt och för att en pipetteringsrobot ska kunna användas för att föra över extrakt till PCR-plattor i ett senare skede utan att hindras av bomullen. Genom att samla ihop och kombinera tidigare resultat kring optimal spårsäkring och extraktionsprotokoll, samt undersöka olika extraktionsrör och tops, var syftet med detta arbete att ta fram ett förbättrat och förenklat protokoll för tops.
I vissa tester inkluderades salivspädningar ner till 1:40 och 1:80. Detta motsvarar en salivmängd på 0,5 µl och 0,25 µl, vilket är mycket lite. Då autolys A-rör ställs mot mikrofugrör (thermoskakprotokoll) är det tydligt att autolys A-rör är
effektivare både för lägre och högre salivmängder. Detta beror på att extraktion med autolys A-rör möjliggör en volymreducering till 200 µl lyseringsbuffert, medan extraktion med mikrofugrör kräver att bomullen är helt täckt (ca 600 µl). Den högre volymen leder till att DNA:t späds och koncentrationen därmed minskar. Eftersom syftet är att eventuellt implementera metoderna på NFC, utfördes standardprotokollet (referensmetoden) precis som det skulle gjorts på NFC. Anledningen till att det är så viktigt att bomullen täcks väl med vätska är att laddningen av qPCR-brunnarna sker automatiskt och pipetteringsroboten som sköter detta går ner med pipettspetsen ca 2 mm under vätskeytan i mitten av röret.
22
För att undvika kollision med bomullen, då denna kan hindra vätskan från att pipetteras upp eller resultera i överföring av chelexkulor som kan hämma PCR-analysen, tillsätts ca 600 µl lyseringsbuffert. Vid användning av rör-i-rörsystem såsom autolys- eller Qiagenrör separeras bomullen från extraktet i slutet av extraktionsprotokollet, och därmed kringgås behovet av större volym vätska. I försöket där tre olika topsar extraherades med SDS-protokoll i autolys A-rör respektive NFC-protokoll i mikrofugrör, observerades en stor skillnad för selefatopsen men inte för de andra två topsen. SDS-protokollet med autolysrör gav högre utbyte för Selefatopsen, medan NFC-protokollet gav lägre DNA-koncentration jämfört med övriga tops. Detta beror troligtvis på att då
Selefatopsen är klippt på standardsätt ”fluffar” bomullen upp sig och suger upp mycket av vätskan, varpå bomullen ”fastnar” i röret och rör sig betydligt mindre under vortex-stegen. Effekten av denna mekaniska kraft minskar därmed och då krävs det en starkare detergent som SDS för en effektiv lysering/frisättning av DNA från bomullen. Det är tydligt att SDS tappar sin fördel mot TWEEN 20 när topsarna klipps av från pinnen eller i mindre format och behåller sin kompakta form, d.v.s. är mindre fluffiga.
Från ett arbetsmetodiskt perspektiv var Puritantopsen enklare att hantera beträffande klippning. Att klippningen blir enklare kan resultera i att
bomullstopsarna inte varierar lika mycket i storlek och ”fluff” vilket också leder till att lyseringsvolymen kan reduceras och DNA-koncentrationen därmed ökas. För både Puritan- och Selefatops testades också att endast klippa den övre halvan (ÖH) av bomullshuvudet. Klippning på detta sätt har många fördelar vad gäller arbetsmetodik och tid. Selefatopsen kan klippas rakt av precis där träskaftet slutar, medan den övre halvan av Puritantopsen klipps rakt av genom plastpinnen. En klippning på detta sätt möjliggör dessutom en reducering av lyseringsvolymen till under 300 µl vid användning av mikrofugrör, vilket ökar DNA-koncentrationen. Då denna studie har utförts systematiskt med minimala variationer i
spårsäkringsteknik, är det oklart hur en sådan klippning kommer stå sig mot verkliga brottsplatsprover, där spårsäkring sker mindre kontrollerat och med variation mellan både spårsäkringsteknik och föremål som topsas. En klippning av övre halvan skulle kräva tydligare riktlinjer för hur spårsäkring ska ske, i syfte att säkra allt DNA på den övre topshalvan. Ett alternativ till att klippa halva
topshuvudet är att använda rör-i rörsystem, tillsammans med antingen hela Puritantopsen eller med Selefa såsom den klipps idag, i kombination med SDS-protokoll istället för NFC-SDS-protokoll. Qiagenröret är det mest lovande av de rör i rörsystem som testats, då autolysrören kräver automatiserad extraktion och iSep-rören visade sig vara otillräckliga ur förvaringssynpunkt då locken inte kunde sluta tätt. Övergång till extraktion med Qiagenrör hade lett till ett enkelt protokoll där extraktionsvätskan kunde standardiserats till 200 µl och filterkorgen,
innehållande bomullen, slängs i slutet. Röret är dessutom anpassat för manuell hantering till skillnad mot autolysrören. Ytterligare optimering av protokollet skulle kunna utföras där övre halvan av topsen klipps i Qiagenrör så att
lyseringsvolymen kan reduceras ytterligare. En stor förbättring kommer troligtvis då till följd av den reducerade volymen chelexlösning som möjliggörs med mindre mängd bomull i röret (Figur 11). På så sätt erhålls ett extrakt med mer koncentrerat DNA. Resultaten från Figur 12 visar även att extraktion med SDS-protokoll generellt ger högre DNA-utbyte jämfört med NFC-SDS-protokollet.
Resultatet från STR-typningen i försöket utfört på NFC stämde väl överens med resultatet från kvantifieringen. De prov som uppvisade hämning i kvantifieringen
23
genererade lägst topphöjder och hade sammanlagt flest antal obalanserade markörer (tabell 2). De fem protokoll som gav högst DNA-koncentration i kvantifieringen gav också de fem högsta topphöjderna, i ordning, vilket indikerar att det inte förekommer någon hämning i amplifieringen. Om det eventuellt skulle finnas hämmande faktorer skulle de framträda tydligare vid typningen än vid kvantifieringen då det till multiplex-PCR tillsätts 17,5 µl extrakt till 7,5 µl PCR-mix. Selefatops, extraherat i iSep-rör med SDS, gav ett högt DNA-utbyte och borde därför ha bland de högsta topphöjderna. Att så inte var fallet beror förmodligen på att det finns någon form av inhibering som trätt fram vid högre extraktkoncentration i multiplex-PCR. Vid extraktion av Selefatops i autolys A-rör med NFC-protokoll blev topphöjden den lägsta medan den lokala balansen mellan topparna hos heterozygota markörer blev högst (närmast 1) bland alla prover.
Iscensatt brottsplats
För den iscensatta brottsplatsen återgavs en högre DNA-koncentration vid
spårsäkring av fingeravtryck med Puritan- än med Selefatops. Eftersom mängden DNA i spåren inte var reglerade kan dock denna skillnad bero på olika mycket DNA i spåret från början. Vid säkring av fingeravtryck med blod klipptes väldigt lite av topsen. Detta beror dels på att hemoglobin i blod har en inhiberande effekt men även att blod innehåller väldigt mycket DNA vilket gör att en mindre del av bomullen räcker. Resten av bomullen extraherades också i ett annat rör för att observera hur utfallet blev. Ett av dessa tre prover erhöll ett väldigt högt Cq-värde vilket indikerar på inhibering, vilket kan förväntas av större mängder blod. Att ett blankt resultat erhölls vid spårsäkring av toppen av pennan ”klicken” kan bero på att det helt enkelt inte fanns tillräckligt med DNA på ytan. Anledningen till att 4N6FLOQSwab inkluderades beror på att en pågående studie har sett positiva utfall vid denna kombinationen.
Metod
Immolase valdes som DNA-polymeras då det i tidigare studier har visat sig vara robust vid närvaro av olika inhibitorer och hämmande bakgrunder, bland annat chelexmatrix [19-20]. För prover som gav PCR-hämning vid användning av Immolase, utfördes qPCR-analysen istället med PerfeCTa qPCR Toughmix, ett kommersiellt kit som har utvecklats för att kunna hantera prover med mycket inhibitorer [19]. Att Toughmix inte fortsättningsvis applicerades för alla prover beror på den stora kostnadsskillnaden mot Immolase, vars mastermix bereds i labbet. Exakt vad Toughmix innehåller är inte känt, men troligtvis består bufferten av olika slags PCR-facilitorer [19]. I så stor utsträckning som möjligt utfördes arbetet med syftet att efterlikna NFC:s rutinmetoder. Alla tops som använts för spårsäkring fick torka i rumstemperatur innan de klipptes, då prover i den verkliga analyskedjan oftast skickas till NFC och har torkat när de klipps. Av samma anledning tillämpas frysningen innan DNA-extraktion, då extraktion inte alltid följer direkt efter spårsäkring.
DNA-extraktioner kan utföras på flera olika sätt. De vanligaste är organisk extraktion, solid fasextraktion och chelexextraktion [21]. Anledningen till att endast chelexextraktioner utfördes var för att det är detta som används på NFC. Extraktion med chelex 100 resin är en snabb, enkel och mycket billig metod som viktigast av allt tillåter extraktionen att ske i ett och samma rör utan några som helst överföringar av vätska och, eventuellt, förlust av DNA. Men eftersom inga
24
reningssteg sker vid Chelex-extraktion, innebär detta ett DNA-extrakt med olika cellkomponenter som eventuellt kan degradera DNA:t vid längre förvaring. Då chelerande molekyler på chelexkulorna binder tvåvärda positiva joner som Mg2+, som i fri form fungerar som kofaktorer för DNaser, skyddas dock DNA:t från nedbrytning [22]. Eventuell närvaro av medföljande PCR-hämmare, som inte heller renas bort, hanteras genom en robust och hämmartålig PCR-kemi, så att amplifiering blir möjlig trots närvaro av hämmare.
PCR-inhibering
Skälet till att de inhiberade proverna kvantifierades med ett annat DNA-polymeras och en annan PCR-mix (toughmix) var att ta reda på om PCR-reaktionen var inhiberad. Vid PCR-inhibering finns det lösningar för att kringgå problemet. Alla olika DNA-polymeraser som finns har olika egenskaper inom värmestabilitet, extensionshastighet, effektivitet, förmåga att inkorporera modifierade baser osv. Dessutom är polymeraser olika känsliga mot PCR-inhibitorer. Exempelvis har inhibering av forensiska DNA-analyser minskat genom att ersätta AmpliTaq Gold med andra alternativa polymeraser som ExTaq Hot Start eller Bio-X-Act Short [23]. För att säkerställa att det var PCR-inhibering som låg bakom orimligt låga kvantifieringsresultat, kvantifierades proverna både spädda och ospädda. Då det förväntade förhållandet mellan Cq-värde för spädda respektive ospädda prover uteblev, kunde slutsatsen att de ospädda proverna orsakade PCR-inhibition dras [15].
I ett försök, ”värmetestet”, undersöktes huruvida inkubering i 100°C bidrar till att plasten i Qiagenröret släpper ifrån sig partiklar som inhiberar qPCR-reaktionen på något sätt. Resultaten visade dock att detta kunde avfärdas. Anledningen till att arbetet med Qiagenröret fortsatte, trots PCR-inhibering vid upprepade tillfällen och få framsteg gällande orsaken till denna inhibering, beror på att det ansågs att inhiberingarna kunde kringgås vid kvantifiering med ett annat, mer robust amplifieringskit, vilket var aktuellt på NFC. Vid senare utfört försök på NFC visade det sig att denna teori stämde, då ingen specifik hämning från Qiagenrören kunde observeras. Inget av de 64 prov som analyserades på NFC gav negativt amplifieringsresultat. För att testa förekomst av inhibitorer innehåller det
kvantifieringskit som används på NFC en intern PCR-kontroll (IPC). IPC består av ett specifikt DNA-templat tillsammans med specifika primers som tillsätts i PCR-mixen. Detta gör att IPC:n bör amplifieras med samma effektivitet (Cq-värde) i alla brunnar som PCR-mix tillsatts till. Om Cq-värdet för IPC:n i ett prov överstiger en definierad övre gräns, indikerar detta att inhibitorer finns närvarande i provet.
KONKLUSION
Det finns många sätt att effektivisera dagens extraktionsmetod för att erhålla högre koncentrationer DNA från forensiska spår. Den mest betydande faktorn för DNA-koncentrationen är volymen av chelexlösningen som tillsätts. För att öka DNA-koncentrationen krävs det en minskad volym vid extraktion. Ett byte från mikrofugrör till ett rör-i-rörsystem ger möjlighet till markant minskad
lyseringsvolym från ca 600 µl till 200 µl. Att byta ut dagens Selefatops mot Puritantops ger en fördel från ett arbetsmetodiskt perspektiv, då topsen är enklare att hantera under klippningen. Dessutom leder Puritantopsen till en mer effektiv
25
lysering, troligtvis till följd av att cellerna/DNA:t frisätts bättre under extraktionen då bomullen håller ihop mer, d.v.s. är mer kompakt. Extrahering av DNA från Puritantops i ett rör-i-rörsystem tillåter extrahering av hela topsen samtidigt som volymen kan pressas ner till 200 µl. Alternativet att kvarstå med mikrofugrör men att minska den klippta bomullsmängden från Selefatopsen (övre halvan) visade sig ha likvärdiga effekter, i synnerhet då SDS användes som detergent istället för Tween20 i chelexlösningen. Det bör dock göras mer systematiska försök för att öka vetskapen om var cellerna sitter på topsen efter en spårsäkring, och om denna klippning lätt kan standardiseras då olika personer utför klippningen.
26
REFERENSER
1. Pestana E A, Belak S, Diallo A, Crowther J R, Viljoen G J. (2010). Early,
Rapid and Sensitive Veterinary Molecular Diagnostics – Real Time PCR Applications. New York.
2. Jeffreys AJ, Wilson V, Thein SL. Individual-specific 'fingerprints' of human DNA. Nature. 1985;316(6023):76-9.
3. Hedman, J. (2011). DNA analysis of PCR-inhibitory forensic samples. Diss. (sammanfattning) Lund, Lunds universitet.
4. Phillips K, McCallum N, Welch L. (2012). A comparison of methods for forensic DNA extraction: Chelex-100® and the QIAGEN DNA Investigator Kit (manual and automated). Forensic Science International: Genetics, 6(2), 282-285.
5. Walsh P S, Metzger D A, Higuchi R. (1991). Chelex 100 as a medium for simple extraction of DNA for PCR-based typing from forensic
material. Biotechniques, 10(4), 506-513.
6. von Wurmb-Schwark N, Mályusz V, Fremdt H, Koch C, Simeoni E, Schwark T. (2006). Fast and simple DNA extraction from saliva and sperm cells obtained from the skin or isolated from swabs. Legal Medicine, 8(3), 177-181.
7. Bio-Rad Laboratories (2000) Chelex® 100 and Chelex 20 Chelating Ion Exchange Resin Instruction Manual.>
http://www.bio-rad.com/webroot/web/pdf/lsr/literature/LIT200.pdf<PDF (2019-03-27)
8. Hedman J, Rådström P. (2013). Overcoming inhibition in real-time
diagnostic PCR. I: Wilks M (Eds.) PCR detection of microbial pathogens (2013). Totowa, NJ. Humana Press,
9. Hedman J, Knutsson R, Ansell R, Rådström P, Rasmusson B. (2013). Pre-PCR processing in bioterrorism preparedness: improved diagnostic
capabilities for laboratory response networks. Biosecurity and bioterrorism:
biodefense strategy, practice, and science, 11(S1), S87-S101.
10. Brown R B, Audet J. (2008). Current techniques for single-cell lysis. Journal
of the Royal Society Interface, 5(suppl_2), S131-S138.
11. Kling L, (2018) Development of a quick and efficient DNA extraction protocol for cigarettes. Master Degree Project in Forensic Science, 30
credits, Uppsala University, Uppsala
12. Hedman J, Knutsson R, Ansell R, Rådström P, Rasmusson B. (2013). Pre-PCR processing in bioterrorism preparedness: improved diagnostic
capabilities for laboratory response networks. Biosecurity and bioterrorism:
27 13. Heid C A, Stevens J, Livak K J, Williams P M. (1996). Real time quantitative
PCR. Genome research, 6(10), 986-994.
14. Didenko V V. (2001). DNA probes using fluorescence resonance energy transfer (FRET): designs and applications. Biotechniques, 31(5), 1106-1121.
15. Schrader A S. (2012). PCR inhibitors – occurrence, properties and removal.
Journal of Applied Microbiology, 113, 1014—1026.
16. Akel Y. (2016) Development of one-tube DNA extraction protocol for crime scene stains. Master Degree Project in Forensic Science, 30 credits, Uppsala
University, Uppsala.
17. Becher C. (2017) Quick and efficient DNA extraction of crime scene traces.
Master Degree Project in Forensic Science, 30 credits, Uppsala University, Uppsala.
18. Hamilton Company, (2019) AutoLyse Tubes (500 tubes). >
https://www.hamiltoncompany.com/automated-liquid-handling/other-robotics/235478<HTML (2019-03-27)
19. Johansson F, (2018) The crucial combination of DNA polymerase, qPCR assay and background matrix. Master thesis 60 credits, Lunds Universitet,
Lund.
20. Sidstedt M, Jansson L, Nilsson E, Noppa L, Forsman M, Rådström P, Hedman J. (2015). Humic substances cause fluorescence inhibition in real-time polymerase chain reaction. Analytical biochemistry, 487, 30-37.
21. Elkins K M, (2013). Forensic DNA Biology. Elsevier.
22. Singh U A, Kumari M, Iyengar S. (2018). Method for improving the quality of genomic DNA obtained from minute quantities of tissue and blood samples using Chelex 100 resin. Biological procedures online, 20(1), 12.
23. Hedman J. Nordgaard A, Rasmusson B, Ansell R, Rådström P. (2009). Improved forensic DNA analysis through the use of alternative DNA
polymerases and statistical modeling of DNA profiles. Biotechniques, 47(5), 951-958.