Möjligheter för fekalt diagnostiskt
test för inflammatory bowel
disease hos hund
Possibilities for faecal diagnostics
for canine inflammatory bowel
disease
Amanda Larsson
Uppsala 2019
Veterinärprogrammet, examensarbete för kandidatexamen, 15 hp
Fakulteten för veterinärmedicin och husdjursvetenskap
Institutionen förbiomedicin och veterinär folkhälsovetenskap
Möjligheter för fekalt diagnostiskt test för
inflammatory bowel disease hos hund
Possibilities for faecal diagnostics for canine
inflammatory bowel disease
Amanda Larsson
Handledare: Erik Pelve, Sveriges lantbruksuniversitet, institutionen för anatomi, fysiologi och biokemi
Examinator: Maria Löfgren, Sveriges lantbruksuniversitet, institutionen för biomedicin och veterinär folkhälsovetenskap
Omfattning: 15 hp
Nivå och fördjupning: Grundnivå, G2E
Kurstitel: Självständigt arbete i veterinärmedicin
Kursansvarig institution: Institutionen för biomedicin och veterinär folkhälsovetenskap Kurskod: EX0862
Program/utbildning: Veterinärprogrammet
Utgivningsort: Uppsala Utgivningsår: 2019
Elektronisk publicering: http://stud.epsilon.slu.se
Nyckelord: IBD, hund, mikrobiom, diagnostiska metoder, fekalt prov, kronisk
tarminflammation
Key words: IBD, dog, canine, microbiome, diagnostic methods, faecal sample, inflammatory
bowel disease
Sveriges lantbruksuniversitet
Swedish University of Agricultural Sciences
Fakulteten för veterinärmedicin och husdjursvetenskap Institutionen för biomedicin och veterinär folkhälsovetenskap
INNEHÅLLSFÖRTECKNING
Sammanfattning ... 1
Summary ... 2
Inledning ... 3
Material och metoder ... 3
Litteraturöversikt ... 3
Det fekala mikrobiomets betydelse i diagnostiken idag och i framtiden ... 3
Mätmetoder ... 4
En beskrivning av QIIME ... 5
Rarefactionanalys ... 5
UniFrac ... 5
Skillnader i mikrobiomet mellan friska hundar och hundar med IBD ... 6
Jämförelse av resultaten i de olika studierna om skillnaderna i mikrobiomet hos hundar med IBD och friska hundar ... 8
Skillnader i mikrobiomet mellan hundar med IBD och hundar med differentialdiagnoser till IBD ... 9
Diskussion ... 10
Litteraturförteckning ... 13
1
SAMMANFATTNING
IBD är en förkortning för inflammatory bowel disease vilket innebär att patienten har inflammation i tarmen och kan få gastrointestinala symtom såsom kräkningar, diarré och illamående. Idag finns inga snabba och enkla diagnostiska metoder för IBD. Hundar med sjukdomen behöver gå igenom många olika undersökningar, däribland gastro- och koloskopi. Den här litteraturstudien undersöker möjligheten till diagnosticering med hjälp av fekal provtagning. Om detta skulle fungera skulle det bidra till mindre lidande för patienterna då de slipper mer invasiva ingrepp. Det är också troligt att de skulle kunna få sin diagnos fortare och därigenom få vänta kortare tid på behandling eftersom färre undersökningar behövs.
Hundar med IBD har en tarmflora som skiljer sig från friska hundars. Det innebär att det, om skillnaderna är tillräckliga, skulle kunna avgöras om en hund har IBD bara genom en analys av mikrobiomet via det fekala provet. Det skulle eventuellt även kunna avgöras om en hund har risk att utveckla IBD genom att undersöka mikrobiomet hos hunden, men detta beror på om det är den störda bakteriefloran som ger IBD eller om det är inflammationen som ger det onormala mikrobiomet.
Resultatet i den här studien är splittrat. Alla artiklar är enade kring att hundar med IBD har en ökad förekomst av Proteobacteria medan artiklarnas resultat är varierande i frågan om Firmicutes då vissa studier påvisar en minskad förekomst och andra en ökad förekomst. De flesta, men inte alla, studier är överens om att fylumet Bacteroidetes minskar hos hundar med IBD.
Mikrobiomet hos hundar med IBD verkar skilja från mikrobiomet hos hundar med andra gastrointestinala sjukdomar, men resultatet är mer tydligt på lägre fylogenetiska nivåer än på fylumnivå. Framförallt är skillnaderna mellan exokrin pankreasinsufficiens och IBD lite oklara eftersom det vid exokrin pankreasinsufficiens är både en ökning av vissa bakterier inom fylumet Firmicutes och en minskning av andra bakterier inom samma fylum och man i forskningen om IBD har tvetydiga resultat vad gäller just detta fylumet Firmicutes där vissa studier ser en ökning och andra en minskning av bakterier inom detta fylum. Här ses en del skillnader lite tydligare på familj-nivå då det ses att det är olika familjer inblandade inom fylumet Firmicutes för de olika sjukdomarna.
Resultatet är överlag lite tvetydigt. Studierna är överens om att mikrobiomen skiljer sig mellan hundar med IBD och friska hundar, så ett diagnostiskt test som bygger på detta bör vara möjligt att utveckla. Det behövs dock mer forskning för att säkerställa exakt på vilka sätt mikrobiomen skiljer sig från varandra.
Fekala test för analys av mikrobiom för att upptäcka sjukdomen IBD är under utveckling inom humanvården och det är med andra ord möjligt att utveckla sådana tester. Tekniken finns idag, det som behövs är ett mikrobiom som kan anses vara standard som sjuka hundars mikrobiom kan jämföras med för att bedöma om de har obalans i mikrobiomet, vilket kallas dysbios. Forskningen behöver fokusera på att hitta bakterier som kan fungera som biomarkörer för att fortsätta utveckla dessa test.
2
SUMMARY
IBD, which is short for inflammatory bowel disease, is a disease which gives the patient gastrointestinal symptoms, such as nausea, diarrhoea and vomiting. Today we do not have a quick, easy test for inflammatory bowel disease. To give the diagnosis IBD, you will have to do multiple examinations of different kinds. If there are an easier way, which is not causing the dog the suffering of having to experience gastro- or coloscopy and that may make it possible to set the diagnosis in a shorter amount of time, that would be beneficial for the patients. This essay is a literature study which investigate the possibility of using faecal samples as a diagnostic method to identify IBD in dogs.
The thought is that the microbial environment in the intestine is different in dogs with this disease than in healthy dogs and that this difference could be a way to distinguish the IBD dogs from dogs with other gastrointestinal disorders. An expansion of that question is the query if it would be possible to detect any indication of the risk of developing the disease, prior to any gastrointestinal symptoms, by doing an analysis of a dog’s microbiome. Whether or not that would be possible depend on if it is the bacteria that causes the inflammation or the inflammation that favour certain kind of bacteria.
The result of this study is divided. In some cases, all studies show clear differences in the composition of bacterial phyla. The phylum of Proteobacteria appears in greater amounts in the IBD dogs than in the healthy control dogs. In that all the studies agree, whilst the results about the phylum Firmicutes are more diffuse with varied results among the articles. Most of the studies, but not all of them, agree that the amount of the phylum Bacteroidetes is decreased in dogs with IBD.
The microbiome in dogs with IBD seems to be different from the microbiome in dogs with other gastrointestinal disorders, but the divergence is clearer on lower phylogenetic levels than on a phylum level. Above all the differences of exocrine pancreatic insufficiency and IBD are uncertain since there is both a increase and a decrease of bacteria in the phylum Firmicutes in the case of exocrine pancreatic insufficiency and the studies of IBD also shows a varied result with both increases and decreases of the phylum. There are in this comparison more differences on a family level than on a phylum level, since the diverseness is seen in different families in the two compared diseases. More studies are needed in this area to investigate more distinct diverseness that can be used in an ulterior diagnostic test.
The kind of diagnostic tests for IBD where you use a faecal sample and analysing the microbiome is under development in the human medicine research. It is in other words possible, but to develop it for dogs there is a need for more research on the standard microbiota in dogs as well as appropriate biomarkers in form of bacteria. The result of this essay is therefore that it would be possible to do this kind of test but in order to develop a reliable diagnostic method there are a need for more studies.
3
INLEDNING
IBD är en förkortning av inflammatory bowel disease, vilket är en kronisk tarmsjukdom som orsakar lidande för de hundar som är drabbade. IBD är en uteslutningsdiagnos och diagnosen ges inte till en hund förrän andra möjliga etiologier till hundens symtom har uteslutits (Simpson & Jergens, 2011).
Vid IBD kan olika symtom från magtarmkanalen uppträda beroende på vilken del av organsystemet som är drabbat. Patienten kan exempelvis ha kroniska kräkningar, diarré och viktminskning. När en utredning av hunden görs är utgångspunkten att undersöka möjliga etiologier till de symtom som hunden uppvisar. Differentialdiagnoserna för symtomet ska därmed uteslutas. Detta kan göras med hjälp av avföringsprover, blodprover och bilddiagnostik. Differentialdiagnoser kan exempelvis vara parasitära infektioner, nybildningar i magtarmkanalen och exokrin pankreasinsufficiens. När andra etiologier har uteslutits genomförs en gastro- eller koloskopi där en biopsi av tarmslemhinnan tas. Biopsin undersöks histologiskt för att utreda om det finns en inflammation i tarmen. (Simpson & Jergens, 2011) Syftet med den här uppsatsen är att besvara om och hur mikrobiomet hos hundar med IBD skiljer sig från mikrobiomet hos friska hundar för att kunna besvara följande frågeställningar: • Är det möjligt att diagnosticera IBD hos hund med hjälp av analys av mikrobiomet i ett
fekalt prov?
• Skulle en analys av mikrobiomet hos en hund kunna ge en indikation för risken att drabbas av IBD?
Ett sådant test skulle vara viktigt för att minska hundarnas lidande vid de diagnostiska undersökningarna eftersom de nuvarande metoderna, gastro- och koloskopi, är invasiva ingrepp i jämförelse med ett enkelt avföringsprov. Det finns framgångsrik forskning på denna typ av diagnostiska tester på människa (Casén et al., 2015), vilket indikerar på att det vore möjligt att utveckla detta även för hund.
MATERIAL OCH METODER
Uppsatsen är en litteraturstudie och de sökord som använts är (dog OR dogs OR canine) AND (microbiome OR microbiota OR gut flora) AND (IBD OR inflammatory bowel disease OR chronic enteritis OR enteropathy). Även sökorden (exocrine pancreatic insufficiency) AND (microbiome OR microbiota OR gut flora) AND (dog) användes samt sökningen (QIIME) AND (microbial) AND (16S rRNA) gjordes. Ännu en sökning med sökorden (microbiome) AND (dog OR canine) AND (diagnosis OR diagnostic test) samt sökningen (microbiome) AND (fecal) AND (diagnosis OR diagnostic test) utfördes. Databaserna Web of Science, PubMed och Google Scholar användes. Även artiklarnas referenser nyttjades samt hemsidor för företag vars produkter tas upp.
LITTERATURÖVERSIKT
Det fekala mikrobiomets betydelse i diagnostiken idag och i framtiden
Idag används inte analys av mikrobiom från fekala prov som ett diagnostiskt test. Det arbetas dock aktivt för att driva fram denna typ av tester inom humanmedicinen. Exempelvis gjordes en studie av Casén et al. (2015) där syftet med undersökningen var att utveckla ett diagnostiskt
4
test för att kunna identifiera dysbios, obalans i mikrobiomet, hos patienter med IBD och IBS (irritable bowel syndrome). Detta skulle då göras med hjälp av analys av mikrobiomet som fåtts fram från ett fekalt prov. I studien deltog 165 friska personer som referensgrupp för att kunna skapa en profil av en frisk tarmflora och genom detta skapades ett dysbiosindex för att kunna bedöma patienters mikrobiom. Därefter undersöktes prov från 46 andra friska personer för att finjustera värdena som fåtts fram samt gjordes det ett test på 127 personer med IBS för att jämföra värdena och kunna skapa modeller för mätmetoden och sätta referensvärden. Profilen som skapades samt indexet användes sedan som utgångspunkt när 287 andra personer provtogs, varav det var en blandning av IBD- och IBS-patienter samt friska frivilliga, för att bedöma metodens kliniska duglighet i att upptäcka dysbios hos patienterna. Resultatet av studien var att dysbios upptäcktes hos 74% av patienterna med IBS, 70% av aldrig behandlade IBD-patienter och 80% av IBD-patienter med avstannad behandling. Hos de friska patienterna hittades dysbios endast hos 16%. De största skillnaderna hos patienterna med IBD i studien jämfört med referensvärdet var en skillnad i förekomsten av Proteobacteria, Firmicutes och Bacteroidetes. Det sågs en ökning av Proteobacteria och en minskning av Firmicutes och Bacteroidetes hos personerna med IBD. Testet beskrivs i studien vara det första kliniska testet som riktar in sig på att upptäcka dysbios med hjälp av fekala prover. Det bedöms dock att fler studier och mer klinisk erfarenhet av testet krävs innan en bedömning av testets tillämplighet kan göras eftersom det förväntas finnas naturliga geografiska skillnader i mikrobiom som inte tagits tillräcklig hänsyn till i denna studie (Casén et al., 2015). Det finns även en annan studie som undersöker möjligheten till fekalt prov för analys av mikrobiom för att diagnosticera IBD hos barn (Papa
et al., 2012). Även de hade resultat som ses som framgångsrika. Dock uttrycker de att metoden
i dagsläget inte kan ersätta gastro- och koloskopi med histologisk analys, men de tycker att den är ett effektivt komplement i diagnosticeringen av barnpatienter med IBD eftersom invasiva ingrepp som gastro- och koloskopi gärna undviks när det handlar om barn (Papa et al., 2012). Liang et al. (2017) har gjort en studie som undersöker om fekala bakterier kan användas som biomarkörer för kolorektalcancer. De har i studien upptäckt flera bakterier som kan associeras med kolorektalcancer och i testet de undersökte i studien hade de tagit fram fyra markörbakterier som skulle ge diagnosen kolorektalcancer. Denna studie tog fram biomarkörer på artnivå och de bakterier som togs fram var Fusobacterium nucleatum, Bacteroides clarus,
Roseburia intestinalis och Clostridium hathewayi. Vidare studier behövs dock även i denna
undersökning då den endast är utformad och testad i en världsdel och mikrobiomet skulle kunna skilja sig på andra kontinenter (Liang et al., 2017).
Enligt en reviewartikel av Suchodolski (2016) finns numera etablerade molekylära metoder för att fastställa dysbios hos hund, men att dessa metoder främst är tillgängliga att användas inom forskning och inte som rutinundersökningar på klinik. Mer kunskap behövs dock för att veta hur informationen som kan utvinnas från dessa metoder ska användas. Idag finns exempelvis inte vetskapen kring behandlingar i förhållande till mikrobiom. (Suchodolski, 2016)
Mätmetoder
I alla studier extraherade man först DNA ur proverna och sekvenserade detta. Därefter gjordes en polymerase chain reaction (PCR) (Van House et al., 2008; Suchodolski et al., 2010, 2012; Isaiah et al., 2017; Omori et al., 2017). PCR är en metod som amplifierar gensekvensen. Detta sker genom att primers, DNA-polymeras och nukleotider tillsätts i DNA-provet. Primern fäster
5
vid en särskild punkt på DNA som sedan med hjälp av DNA-polymeraset och nukleotiderna replikeras (PCR-metoden - SVA). Sekvenserna som extraherats analyseras på olika sätt. Analyser som görs är att jämföra 16S rRNA med data i olika databaser och använda sig av olika mjukvaror till en dator som utför analyser. Mjukvaror som använts för analys av gensekvenser är QIIME, B2C2, PRIMER 6, CLUSTAL_W, BIOEDIT, DOTUR, CHECK_CHIMERA, CHROMASPRO, PICRUSt och PINTAIL (Van House et al., 2008; Suchodolski et al., 2010, 2012b; Isaiah et al., 2017; Omori et al., 2017). QIIME användes för att skapa rarefactionkurvor, vilket illustrerar en uppskattning av mångfalden av bakterier i ett prov (Suchodolski et al., 2012b; Isaiah et al., 2017; Kim et al., 2017; Omori et al., 2017). Nämnda mjukvaroprogram kan också ta fram ett UniFrac-värde, vilket är ett sätt att hantera genetiska data för att kunna jämföra olika mikrobiom med varandra (Van House et al., 2008; Suchodolski et al., 2010, 2012b; Isaiah et al., 2017; Omori et al., 2017).
En beskrivning av QIIME
I den här litteraturstudien ges en beskrivning av mjukvaran QIIME eftersom den är den mest frekvent använda mjukvaran av de som nämnts i studerade artiklar (Suchodolski et al., 2012b; Isaiah et al., 2017; Omori et al., 2017). QIIME är ett program skapat för att analysera mikrobiom från extraherade DNA-sekvenser (Hall & Beiko, 2018). QIIME står för Quantitative Insights Into Microbial Ecology och används för att analysera 16S rRNA, som är en markörgen för taxonomisk tillhörighet. Denna tas fram genom att isolera särskilda gener, fylogenetiskt informativa gener, vilka sedan amplifieras med hjälp av PCR. I genen finns variationer som används för att identifiera de olika taxonomiska grupperna och genom att mäta antalet variationer kan bakteriernas proportioner till varandra samt mångfalden av organismer i provet uppskattas. För att göra detta använder sig programmet av bioinformatiska metoder samt jämförelser med databaser. För att programmet ska kunna mäta diversiteten i ett prov måste ett fylogenetiskt träd byggas upp. QIIME kan sedan utifrån detta ge ett UniFrac-värde. QIIME kan också utföra rarefactionanalyser (Hall & Beiko, 2018).
Rarefactionanalys
QIIME kan, som nämnts ovan, utföra en alfa-rarefactionanalys för att undersöka att analysen av mikrobiomet i ett prov har varit tillräckligt djupgående för att inte ha missat ett taxa. Om analysen är tillräckligt djupgående innebär det att tillräckligt många avläsningar har gjorts på provet. Av denna rarefaction kan programmet skapa en kurva som baseras på något som kallas Shannon Diversity (Hall & Beiko, 2018). Shannon Diversity är ett diversitetsindex som beräknar information kring släktskap och diversitet inom ett bakteriesamhälle. Detta beräknas med hjälp av det totala antalet mikroorganismer i samhället, proportionen av den efterfrågade bakterien samt en siffra på hur jämnt bakterien var fördelad i provet. Rarefaction är en statistisk metod som ska uppskatta hur många operativa taxonomiska enheter (OTU) som finns i ett prov, detta för att mäta mångfalden av bakterier. OTU utläses då med hjälp av DNA-sekvensen och kan avläsas på olika taxonomiska nivåer (Kim et al., 2017).
UniFrac
År 2005 beskrevs en ny metod för datorhantering av genetiska data, som kan visa likheten mellan olika bakteriella miljöer. Metoden kallas för UniFrac och mäter det fylogenetiska avståndet mellan olika taxa i ett fylogenetiskt träd. Avståndet mäts med andra ord mellan ett visst mikrobiom jämfört med ett annat mikrobiom. Innan denna metod utvecklades användes
6
metoder som kunde ge resultat om de mikrobiom som jämfördes var 95% identiska eller mer. I övrigt gjorde dessa metoder ingen skillnad på om de jämförda mikrobiomen var 3% identiska eller 40% identiska. Fördelen med UniFrac är således att mer information kan fås även mellan de bakteriella miljöer som är mindre än 95% identiska. Med hjälp av UniFrac kan graden av skiljaktighet mätas mellan olika gensekvenser. Vid denna metod används 16S rRNA-genen. UniFrac kan användas för att jämföra flera olika bakteriesamhällen samtidigt eftersom det fungerar som en måttenhet och därmed kan jämföras med standardiserade värden. (Lozupone & Knight, 2005)
Skillnader i mikrobiomet mellan friska hundar och hundar med IBD
År 2010 gjordes en studie där det utfördes endoskopi på hundarna för att få fram biopsier varifrån bakterie-DNA kunde extraheras (Suchodolski et al., 2010). I IBD-gruppen fanns sju hundar varav fyra hanar och tre tikar. Medianåldern i gruppen var 8,5 år och alla hundar i gruppen tillhörde olika raser. I kontrollgruppen var det sju hundar och alla var tikar. Medianåldern i kontrollgruppen var 6 år och två var beaglar och resten var blandrashundar. Efter extraktionen av DNA gjordes en amplifiering av 16S rRNA-genen. Klonade gensekvenser jämfördes sedan med tidigare befintliga 16S rRNA-gener med hjälp av bland annat GenBank och olika programvaror användes för att strukturera upp gensekvenserna. Bland annat användes ett program kallat DOTUR för att bestämma operativa taxonomiska enheter, vilka gavs definitionen: mindre än 2% skillnad och 98% likhet mellan sekvenserna. Olika typer av analyser gjordes där gensekvensen jämfördes med de som fanns i databaser samt att metoden UniFrac användes. Signifikanta skillnader hittades med flera av analysmetoderna, däribland UniFrac. De största skillnaderna hittades på genusnivå, men indikationer för skillnader på fylumnivå konstaterades också. Hos hundar med IBD hittades rikligare mängd av bakterier som hör till fylumet Proteobacteria än hos kontrollgruppen. Detta gällde tre varianter av Proteobacteria,
Alfa-, Beta- och Gamma-proteobacteria. Ett exempel inom detta fylum är bakterien Pseudomonas vilken förekom hos alla sju hundar med IBD men bara hos en hund i
kontrollgruppen. En annan inom samma fylum är bakterien Acinetobacter, vilken förekom hos 6 av hundarna med IBD och förekom inte alls hos kontrollgruppen (Suchodolski et al., 2010). Båda dessa bakteriearter tillhör klassen Gammaproteobacteria (VetBact, u.å.a, VetBact, u.å.b). En annan skillnad som påpekades var att hundarna med IBD inte hade någon förekomst av Clostridier jämfört med förekomst hos fyra hundar i kontrollgruppen (Suchodolski et al., 2010). Genus Clostridium tillhör fylumet Firmicutes (VetBact, u.å.c).
I en studie har det, med hjälp av ett t-test, setts att mångfalden av bakteriearter i mikrobiomet var signifikant lägre hos hundar med IBD än hos friska hundar (Van House et al., 2008). Antalet hundar i studiens kontrollgrupp var nio och antalet hundar i IBD-gruppen var tio. Hundarna i IBD-gruppen var av varierande raser, en av varje ras, medan variationen i kontrollgruppen var mindre med bara två representerade raser varav ingen av dessa fanns representerade i IBDgruppen. Könen i IBD-gruppen var jämnt fördelade medan det i kontrollgruppen var en tredjedel hanar och två tredjedelar tikar. Proverna togs via endoskopi. Hundarna med IBD hade en ökad förekomst av bakterier inom familjen Enterobacteriaceae. Det påvisades också att hundarna med IBD hade 14,2 procentenheter fler Firmicutes samt 8,5 procentenheter färre Bacteroidetes. Spirochaetes förekom inte alls hos hundarna med IBD medan de friska hundarnas mikrobiom bestod till 10,3% av Spirochaetes. Majoriteten av de kontrollgruppens tarmflora består av familjerna Clostridiaceae, Carnobacteriaceae och Helicobacteriaceae,
7
medan hundarna med IBD har en tarmflora som främst består av Clostridiaceae,
Enterobacteriaceae och Streptococaceae. Hundarna med IBD hade 20,2 procentenheter fler av
familjen Enterobacteriaceae, som hör till fylumet Proteobacteria (Van House et al., 2008) och klassen gammaproteobacteria (VetBact, u.å.d), och 20,3 procentenheter fler Clostridiaceae, som tillhör fylumet Firmicutes. Proportionellt var det också fler av hundarna med IBD som hade förekomst av Enterobacteriaceae medan det var lika vanligt med förekomst av
Clostridiaceae hos friska hundar som hos hundar med IBD även om mängden var större hos de
sistnämnda. (Van House et al., 2008).
I studierna av Baumgart et al. (2007, Janeczko et al. (2008), Xenoulis et al. (2008) och Suchodolski et al. (2010) som sammanfattas i studien av Simpson & Jergens (2011) påvisas det hos flera olika arter en förskjutning i andelen gramnegativa bakterier respektive grampositiva bakterier i förhållande till varandra i mikrobiomet som kunde associeras med inflammatoriska tillstånd i magtarmkanalen (Simpson & Jergens, 2011). Förskjutningen innebär att grampositiva Firmicutes är färre samtidigt som gramnegativa Proteobacteria, såsom familjen
Enterobacteriaceae, är fler hos individer med inflammation i tarmen (Simpson & Jergens,
2011). Det har exempelvis setts en korrelation mellan höga nivåer av Enterobacteriaceae i mikrobiomet och tarminflammation hos katt (Janeczko et al., 2008). I samma artikel har det också påvisats en korrelation mellan onormal struktur på slemhinnan och graden av infiltration av inflammatoriska celler, såsom makrofager och lymfocyter, och förekomsten av
Enterobacteriaceae och Clostridium spp. Denna studie tog sina mikrobiotiska prover med hjälp
av endoskopi (Janeczko et al., 2008). Hos hundrasen Boxer har det påvisats att en bibehållen förbättring av granulomatös inflammation i tarmen korrelerar med utrotning av invasiva
Escherichia coli och det finns potentiellt ett kausalt samband (Mansfield et al., 2009). Escherichia coli tillhör familjen Enterobacteriaceae, familjen Gammaproteobacteria och
fylumet Proteobacteria (VetBact, u.å.d). Escherichia coli är en kommensal i tarmen, men vissa typer av bakterien kan orsaka tarmsjukdom som kan ge hunden diarré. Escherichia coli kan också ge urinvägsinfektion hos hund (Escherichia coli - SVA; Beutin, 1999).
I en nyare studie från 2017 påvisades, till skillnad från i de tidigare studierna, inga signifikanta skillnader mellan hundar med IBD och kontrollgruppen på fylumnivå. Grupperna i denna studie hade lika många tikar som hanar i IBD-gruppen och en hane mer än antalet tikar i kontrollgruppen. Genomsnittsåldern i dessa två grupper var likvärdig och raserna hade stor variation i båda grupperna, ungefär en individ per ras, och samma raser fanns representerade i båda grupperna. Antalet individer i kontrollgruppen var 15 stycken och i IBD-gruppen åtta stycken. I denna studie användes avföringsprover för att utföra undersökningen (Omori et al., 2017). I en annan undersökning från 2014 (Honneffer et al., 2014), som gjorts som en litteraturstudie, visas det på att det finns dysbios hos hundar med IBD och den förskjutning som setts hos hund är en minskning av fylumen Firmicutes och Bacteroidetes i kombination med en ökning av fylumen Proteobacteria och Actinobacteria och dessutom i även en ökning av ovan nämnda Bacteroidetes. Ökningen av de sist nämnda bakterierna samt minskningen av Bacteroidetes har påvisats med hjälp av prover från slemhinnan, medan minskningen av Firmicutes har påvisats både med föregående metod samt med fekala prover. De här resultaten är sammanfattade i en studie av Honneffer et al. (2014) och resultaten kommer ifrån början från studierna Xenoulis et al. (2008), Suchodolski et al. (2010) samt Suchodolski et al. (2012a). I undersökningen från 2017 (Omori et al.) kunde dessa förskjutningar som visades 2014 inte påvisas med signifikans. Däremot kunde signifikanta skillnader visas på lägre fylogenetiska
8
nivåer. Familjen Paraprevotellaceae, inom fylumet Bacteroidetes, förekom med 0,05 procentenheter högre antal hos hundar med IBD. Den tidigare omnämnda skillnaden i förekomst av Firmicutes kunde inte påvisas med signifikans i den här studien däremot sågs att Eubacteriaceae (Omori et al., 2017), som tillhör fylum Firmicutes (Eubacteriaceae, 2018), förekom i större mängder hos hundar med IBD. Precis som i studien från 2010 användes UniFrac för att mäta skillnaderna mellan mikrobiomen (Omori et al., 2017).
En annan studie tog avföringsprover på totalt 19 hundar med IBD varav 10 stod på behandling och 9 var obehandlade och nyss diagnosticerade. Kontrollgruppen bestod av 32 hundar. Båda grupperna bestod av privatägda hundar som bodde i hemmiljö. I denna studie sågs inga signifikanta skillnader mellan hundar med IBD och kontrollgruppen. I studien kunde en trend ses att hundar med obehandlad, aktiv IBD hade en minskning av Faecalibacterium spp. och
Turicibacter spp. jämfört med både kontrollgruppen samt jämfört med gruppen med behandlad,
inaktiv IBD (Suchodolski et al., 2012b). Arten Faecalibacterium spp. tillhör fylumet Firmicutes (Faecalibacterium, 2019) vilket även Turicibacter spp. gör (Turicibacter, 2018). I studien kunde en signifikant minskning av genuset Fusobacterium ses hos hundar med obehandlad, aktiv IBD jämfört med kontrollgruppen och en signifikant ökning av bakterien kunde ses under perioder då hundarna inte hade kliniska symtom på IBD jämfört med perioden då symtom visade sig (Suchodolski et al., 2012b). Genuset Fusobacterium hör till fylumet Fusobacteria (VetBact,
u.å.e).
Jämförelse av resultaten i de olika studierna om skillnaderna i mikrobiomet hos hundar med IBD och friska hundar
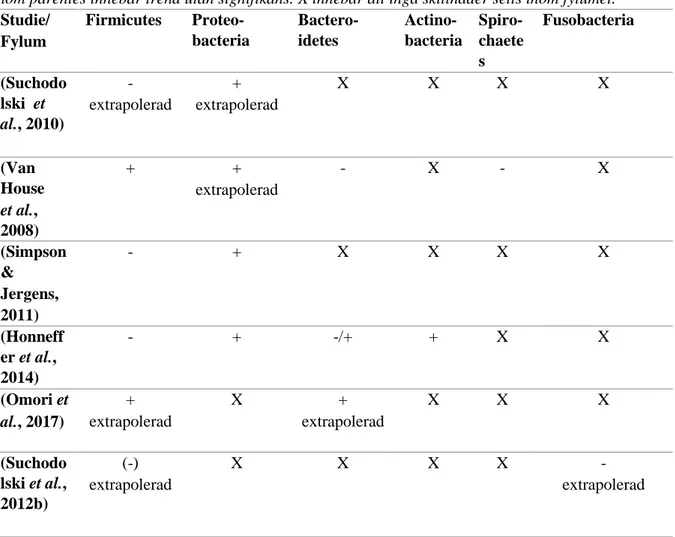
Alla studier ser en ökad förekomst av Proteobacteria hos hundar med IBD jämfört med friska hundar (Tabell 1). I studien av Suchodolski et al. (2010) nämns att det är just alfa-, beta- och
gamma-proteobaceria, i andra studier, såsom studierna av Van House et al. (2008) och Simpson
and Jergens, (2011), nämns framförallt familjen Enterobacterieaceae, vilken hör till klassen
gammaproteobacteria inom fylumet Proteobacteria.
Om vi istället tittar på studiernas resultat av fylumet Firmicutes, är det inte lika tydligt (Tabell 1). I studien från Van House et al. (2008) är resultatet att vi har 14,2 procentenheter fler Firmicutes hos hundar med IBD än hos kontrollgruppen. I studierna från Simpson & Jergens (2011) samt från Honneffer et al. (2014) skrivs det istället om att fylumet Firmicutes förekommer i mindre grad hos hundar med IBD. De sist nämnda studierna är dock inte studier som påvisar ett eget resultat utan de gör en studie av andras forskning. I studien av Suchodolski
et al. från 2010 stödjer dock resultatet dessa studiers resultat i och med att inga bakterier inom
genuset Clostridium, vilket tillhör fylumet Firmicutes, hade påvisats hos hundarna med IBD jämfört med 4 stycken i kontrollgruppen. I studien av Omori et al. (2017) kunde ingen signifikant skillnad av detta fylum påvisas, däremot sågs en signifikant ökning av familjen Eubacteriaceae, vilken tillhör fylumet Firmicutes. I den andra studien av Suchodolski et al. från 2012, kunde inga signifikanta skillnader vad gäller Firmicutes kunde påvisas, dock kunde en trend som visade på minskade nivåer av fylumet ses. Detta är med andra ord ett mindre tydligt resultat än det om Proteobacteria.
Resultaten för fylumet Bacteroidetes också ganska tvetydligt (Tabell 1). I studien från (Van House et al., 2008) är resultatet att hundar med IBD har 8,5 procentenheter färre av fylumet
9
Bacteroidetes än kontrollgruppen. I studien från Honneffer et al. (2014) skrivs att det finns både en minskning och en ökning av förekomsten av bakterier inom fylumet Bacteroidetes hos hundar med IBD jämfört med friska hundar. Studien från Omori et al. (2017) påvisade en signifikant ökning med 0,05 procentenheter av familjen Paraprevotallaceae, vilken tillhör fylumet Bacteroidetes, hos hundar med IBD jämfört med kontrollgruppen. Därav kan vi inte heller där dra säkra slutsatser kring hur skillnaden mellan hundar med IBD och friska hundar ser ut.
Studien av Suchodolski et al. från 2012 påvisade en signifikant ökning av genuset Fusobacterium hos hundar med aktiv IBD. Denna skillnad i mikrobiom har inte någon av de andra studierna påtalat. Studien av Honneffer et al. (2014) påvisar en ökning av Actinobacteria, vilket heller ingen annan studie påvisat. (Tabell 1)
Tabell 1.Sammanställning över ökningar och minskningar inom olika fylum i respektive studie. I de fall en skillnad i förekomst setts på lägre fylogenetiska nivåer har detta extrapolerats till fylumnivå. Resultat inom parentes innebär trend utan signifikans. X innebär att inga skillnader setts inom fylumet.
Studie/ Fylum Firmicutes Proteo-bacteria Bactero-idetes Actino-bacteria Spiro-chaete s Fusobacteria (Suchodo lski et al., 2010) -extrapolerad + extrapolerad X X X X (Van House et al., 2008) + + extrapolerad - X - X (Simpson & Jergens, 2011) - + X X X X (Honneff er et al., 2014) - + -/+ + X X (Omori et al., 2017) + extrapolerad X + extrapolerad X X X (Suchodo lski et al., 2012b) (-) extrapolerad X X X X -extrapolerad
Skillnader i mikrobiomet mellan hundar med IBD och hundar med differentialdiagnoser till IBD
En differentialdiagnos till IBD är exokrin pankreasinsufficiens (Simpson & Jergens, 2011). Precis som vid IBD uppstår en bakteriell dysbios hos hundar med exokrin pankreasinsufficiens. Hos dessa hundar ses signifikant högre halter av familjerna Bifidobacteriaceae,
Enterococcaceae och Lactobacillaceae. Bifidobacteriaceae hör till fylumet Actinobacteria
10
samma fylum som föregående (Lactobacillaceae, 2017; Enterococcaceae, 2018). Familjerna
Lachnospiraceae och Ruminococcaceae var istället signifikant färre hos hundarna med exokrin
pankreasinsufficiens (Isaiah et al., 2017). Både Lachnospiraceae och Ruminococcaceae hör till fylumet Firmicutes (Lachnospiraceae, 2018; Ruminococcaceae, 2018).
En annan differentialdiagnos till IBD är lymfom i tarmen. I en studie från 2017 (Omori et al.) framkom det att mikrobiomet mellan hundar med IBD signifikant skiljde sig från mikrobiomet hos hundar med lymfom i tarmen. UniFrac-avståndet skiljde sig signifikant mellan grupperna. Hundar med lymfom i tarmen har 0,14 procentenheter fler Parabacterioides, tillhörande fylum Bacteroidetes, än både friska hundar och hundar med IBD (Omori et al., 2017).
DISKUSSION
Alla studier har kommit fram till att det finns skillnader i mikrobiomet mellan hundar med IBD och friska hundar. En skillnad som återfunnits i samtliga studier som nämnts i litteraturöversikten är att hundar med IBD har en ökad förekomst av Proteobacteria.
I en studie från Janeczko et al. (2008) sågs det en korrelation mellan en onormal struktur på slemhinnan samt infiltration av inflammatoriska celler och förekomsten av Enterobacteriaceae spp., fylum Proteobacteria, och Clostridium spp., fylum Firmicutes. Detta ger frågan om det är bakterierna som skapar en förändrad struktur på slemhinnan eller om det är den onormala strukturen som gynnar just dessa bakteriers tillväxt. Denna fråga anser jag vara intressant då denna till viss del skulle kunna besvara frågeställningen kring om risken för IBD skulle kunna förutsägas hos en hund med hjälp av fekalt prov. Om det är bakterierna som skapar den förändrade strukturen borde det vara möjligt att se detta i ett avföringsprov, medan om det är inflammationen i sig som skapar denna onormalitet och ger ökad tillväxt av dessa bakterier ses detta på ett avföringsprov inte förrän IBD hos hunden redan är kliniskt. I studien av Mansfield
et al. (2009) mår hundarna bättre efter att ha fått behandling mot invasiva Escherichia coli.
Dessa tillhör Proteobacteria och att de blev bättre tänker jag ger en indikation på att det är bakterierna som orsakat inflammationen istället för tvärtom.
Studierna har använt sig av olika provtagningsmetoder, vissa har använt sig av feces och andra av biopsier från tarmslemhinnan. Detta kan möjligtvis påverka resultatet eftersom alla bakterier som finns i tarmen inte nödvändigtvis utsöndras via avföringen medan de prover som tas med biopsi får ett resultat som stämmer bättre överens med hur det faktiskt ser ut i tarmen. Denna uppsats utgår dock från frågeställningen om IBD skulle kunna diagnosticeras enbart med analys av patientens feces. I detta fall blir då de resultat som grundats på just feces de som är mest relevanta. Resultaten mellan studier som använts sig av fekala prover skiljer sig inte markant från de som använt sig av biopsier så kanske har provtagningsmetoden inte enorm betydelse även om den ska tas i beaktande.
Vissa av studierna hade en större mångfald av raser i sina studiegrupper vilket jag tänker är positivt då bilden bör bli mer generell angående arten hund och inte bara en viss ras. Andra studier hade bra mångfald i sin IBD-grupp men desto färre representerade raser i kontrollgruppen. Detta beror kanske på att vissa hundraser mer frekvent används som försöksdjur, men det skulle kunna påverka referenspunkten i jämförelsen av mikrobiomet mellan grupperna då mångfalden är mindre.
11
Om vi tittar på avsnittet kring differentialdiagnoser kan vi se att en ökning av Firmicutes och Actinobacteria kan ses i mikrobiomet hos hundar med sjukdomen exokrin pankreasinsufficiens. Det ses dock även en minskning av mängden bakterier av andra arter inom fylumet Firmicutes. Det innebär att det vid diagnostiska avföringsprov behöver gås ner på lägre fylogenetiska nivåer för större säkerhet. Jämförs detta med mikrobiomet hos hundar med IBD ses att det även skulle kunna innebära en osäkerhet i diagnosen baserat på dessa bakterier om fekalt prov tas. Detta eftersom det finns en studie på ökade mängder Firmicutes samt en studie som påvisar en ökning i mängden Actinobacteria hos hundar med IBD. Angående Firmicutes finns det dock flera andra studier som påvisar motsatsen, med andra ord en minskning av Firmicutes hos hundar med IBD. På lägre fylogenetiska nivåer ses att det i fallen med IBD främst påvisats skillnader i familjen
Clostridiaceae vilket skiljer sig från de som omnämnts i artikeln om exokrin
pankreasinsufficiens, det vill säga familjerna Bifidobacteriaceae, Enterococcaceae,
Lactobacillaceae, Lachnospiraceae och Ruminococcaceae. Fler studier behöver göras för att
kunna säkerställa om dessa skillnader är tillräckliga för att kunna skilja sjukdomarna åt bara genom ett fekalt prov.
Hundar med lymfom i tarmen har enligt studien av Omori et al. (2017) signifikanta skillnader i mikrobiomet jämfört med hundar med IBD. Detta har påvisats med UniFrac, vilket bör kunna användas vid diagnostiska analysen av avföringsproverna för att skilja sjukdomarna åt. Bland annat kunde det ses att hundar med lymfom i tarmen hade 0,14 procentenheter fler
Parabacterioides än hundar med IBD och hundarna i kontrollgruppen. Parabacterioides tillhör
fylumet Bacteroidetes och flera av studierna som finns i litteraturöversikten är eniga i att Bacteroidetes hos hundar med IBD har lägre förekomst än hos friska hundar. Med andra ord bör detta kunna ses som relativt säkert eftersom hundar med lymfom i tarmen har en ökning av Bacteroidetes medan hundar med IBD generellt har en minskning av samma fylum. Dock vore fler studier för att säkerställa minskningen av Bacteroidetes hos hundar med IBD på sin plats för att ett eventuellt diagnostiskt prov skulle vara helt tillförlitligt.
Frågan är då om det är möjligt att skapa ett diagnostiskt test för IBD hos hund som bygger på analys av mikrobiomet i ett fekalt prov. Med tanke på de studier som gjorts på människa där framställningen av ett sådant prov lyckats, även om det inte är helt klart ännu bör det vara möjligt även för hund. Vi är inte där ännu. Det behövs fler studier för att kunna få fram de bakterier som hos hund kan fungera som biomarkörer för sjukdomen. Studien av Casén et al. (2015) ger tanken att de studier som behövs för att få fram biomarkörs-bakterier också måste generaliseras genom att göra dessa på hundar på olika platser i världen, kanske med olika diet och generellt olika förutsättningar, för att testet som utvecklas ska vara så allomfattande och täckande för alla hundar som möjligt.
Suchodolski (2016) nämner i sin reviewartikel att det idag är möjligt att upptäcka en dysbios hos hund, även om detta enbart används inom forskning. Dysbios innebär som tidigare skrivits en obalans i mikrobiomet. Detta kan uppkomma utan att patienten har just IBD. Så egentligen finns metoden för att upptäcka denna obalans i mikrobiomet redan, det viktiga är då att kunna skilja den dysbios som uppstår vid IBD från dysbios som uppstår av andra orsaker eller andra sjukdomar. Därav att rätt biomarkörer behöver studeras fram.
Slutsatsen är att det finns alla möjligheter att utveckla ett diagnostiskt test, som bygger på en analys av mikrobiomet i ett fekalt prov, för IBD hos hund eftersom tekniken finns och metoden
12
redan håller på att utvecklas på människa. De studier som finns på vilka bakterier som skiljer sig åt i mikrobiomet hos hundar med IBD och friska hundar är dock inte överens i alla aspekter även om de alla fått ett enhälligt resultat att Proteobacteria är ett fylum som utgör en stor skillnad. Det behövs mer kunskap för att säkert kunna få fram fler bra biomarkörer.
13
LITTERATURFÖRTECKNING
Baumgart, M., Dogan, B., Rishniw, M., Weitzman, G., Bosworth, B., Yantiss, R., Orsi, R.H., Wiedmann, M., McDonough, P., Kim, S.G., Berg, D., Schukken, Y., Scherl, E. & Simpson, K.W. (2007). Culture independent analysis of ileal mucosa reveals a
selective increase in invasive Escherichia coli of novel phylogeny relative to depletion of Clostridiales in Crohn’s disease involving the ileum. The ISME Journal, vol. 1 (5), ss. 403–418.
Beutin, L. (1999). Escherichia coli as a pathogen in dogs and cats. Veterinary Research, vol. 30 (2–3), ss. 285–298.
Bifidobacteriaceae (2018). Wikipedia. Available from:
https://en.wikipedia.org/w/index.php?title=Bifidobacteriaceae&oldid=829738191. [Accessed 2019-03-03].
Casén, C., Vebø, H.C., Sekelja, M., Hegge, F.T., Karlsson, M.K., Ciemniejewska, E., Dzankovic, S., Frøyland, C., Nestestog, R., Engstrand, L., Munkholm, P., Nielsen, O.H., Rogler, G., Simrén, M., Öhman, L., Vatn, M.H. & Rudi, K. (2015). Deviations in human gut microbiota: a novel diagnostic test for determining dysbiosis in patients with IBS or IBD. Alimentary Pharmacology & Therapeutics, vol. 42 (1), ss. 71–83. Enterococcaceae (2018). Wikipedia. Available from:
https://en.wikipedia.org/w/index.php?title=Enterococcaceae&oldid=831608864. [Accessed 2019-04-06].
Escherichia coli - SVA. Available from:
https://www.sva.se/djurhalsa/notkreatur/endemiskasjukdomar-notkreatur/diarre-hos-smakalvar/escherichia-coli-kalv. [Accessed 2019-0406].
Eubacteriaceae (2018). Wikipedia. Available from:
https://en.wikipedia.org/w/index.php?title=Eubacteriaceae&oldid=829768864. [Accessed 2019-03-17].
Faecalibacterium (2019). Wikipedia. Available from:
https://en.wikipedia.org/w/index.php?title=Faecalibacterium&oldid=883698252. [Accessed 2019-03-03].
Hall, M. & Beiko, R.G. (2018). 16S rRNA Gene Analysis with QIIME2. I: Beiko, R.G., Hsiao, W., & Parkinson, J. (red) Microbiome Analysis: Methods and Protocols. New York, NY: Springer New York, ss. 113–129.
Honneffer, J.B., Minamoto, Y. & Suchodolski, J.S. (2014). Microbiota alterations in acute and chronic gastrointestinal inflammation of cats and dogs. World Journal of
Gastroenterology : WJG, vol. 20 (44), ss. 16489–16497.
Isaiah, A., Parambeth, J.C., Steiner, J.M., Lidbury, J.A. & Suchodolski, J.S. (2017). The fecal microbiome of dogs with exocrine pancreatic insufficiency. Anaerobe, vol. 45, ss. 50– 58 (Special issue: Anaerobe 2016).
Janeczko, S., Atwater, D., Bogel, E., Greiter-Wilke, A., Gerold, A., Baumgart, M., Bender, H., McDonough, P.L., McDonough, S.P., Goldstein, R.E. & Simpson, K.W. (2008). The relationship of mucosal bacteria to duodenal histopathology, cytokine mRNA, and clinical disease activity in cats with inflammatory bowel disease. Veterinary
Microbiology, vol. 128 (1), ss. 178–193.
Kim, B.-R., Shin, J., Guevarra, R., Lee, J.H., Kim, D.W., Seol, K.-H., Lee, J.-H., Kim, H.B. & Isaacson, R. (2017). Deciphering Diversity Indices for a Better Understanding of Microbial Communities. Journal of Microbiology and Biotechnology, vol. 27 (12), ss. 2089–2093.
14
https://en.wikipedia.org/w/index.php?title=Lachnospiraceae&oldid=849770741. [Accessed 2019-04-06].
Lactobacillaceae (2017). Wikipedia. Available from:
https://sv.wikipedia.org/w/index.php?title=Lactobacillaceae&oldid=40095542. [Accessed 2019-03-03].
Liang, Q., Chiu, J., Chen, Y., Huang, Y., Higashimori, A., Fang, J., Brim, H., Ashktorab, H., Ng, S.C., Ng, S.S.M., Zheng, S., Chan, F.K.L., Sung, J.J.Y. & Yu, J. (2017). Fecal Bacteria Act as Novel Biomarkers for Noninvasive Diagnosis of Colorectal Cancer.
Clinical Cancer Research, vol. 23 (8), ss. 2061–2070.
Lozupone, C. & Knight, R. (2005). UniFrac: a New Phylogenetic Method for Comparing Microbial Communities. Applied and Environmental Microbiology, vol. 71 (12), ss. 8228–8235.
Mansfield, C.S., James, F.E., Craven, M., Davies, D.R., O’Hara, A.J., Nicholls, P.K., Dogan, B., MacDonough, S.P. & Simpson, K.W. (2009). Remission of Histiocytic Ulcerative Colitis in Boxer Dogs Correlates with Eradication of Invasive Intramucosal
Escherichia coli. Journal of Veterinary Internal Medicine, vol. 23 (5), ss. 964–969. Omori, M., Maeda, S., Igarashi, H., Ohno, K., Sakai, K., Yonezawa, T., Horigome, A.,
Odamaki, T. & Matsuki, N. (2017). Fecal microbiome in dogs with inflammatory bowel disease and intestinal lymphoma. Journal of Veterinary Medical Science, vol. 79 (11), ss. 1840–1847.
Papa, E., Docktor, M., Smillie, C., Weber, S., Preheim, S.P., Gevers, D., Giannoukos, G., Ciulla, D., Tabbaa, D., Ingram, J., Schauer, D.B., Ward, D.V., Korzenik, J.R., Xavier,
R.J., Bousvaros, A. & Alm, E.J. (2012). Non-Invasive Mapping of the Gastrointestinal Microbiota Identifies Children with Inflammatory Bowel Disease. PLOS ONE, vol. 7 (6), s. e39242.
PCR-metoden - SVA. Available from: https://www.sva.se/analyser-och-produkter/pcrmetoden.
[Accessed 2019-04-06].
Ruminococcaceae (2018). Wikipedia. Available from:
https://en.wikipedia.org/w/index.php?title=Ruminococcaceae&oldid=865258265. [Accessed 2019-03-03].
Simpson, K.W. & Jergens, A.E. (2011). Pitfalls and Progress in the Diagnosis and
Management of Canine Inflammatory Bowel Disease. Veterinary Clinics of North
America: Small Animal Practice, vol. 41 (2), ss. 381–398 (Chronic Intestinal Diseases
of Dogs and Cats).
Suchodolski, J.S. (2016). Diagnosis and interpretation of intestinal dysbiosis in dogs and cats.
The Veterinary Journal, vol. 215, ss. 30–37 (Special Issue: Recent Developments in
Veterinary Diagnostics).
Suchodolski, J.S., Dowd, S.E., Wilke, V., Steiner, J.M. & Jergens, A.E. (2012a). 16S rRNA Gene Pyrosequencing Reveals Bacterial Dysbiosis in the Duodenum of Dogs with Idiopathic Inflammatory Bowel Disease. PLOS ONE, vol. 7 (6), s. e39333.
Suchodolski, J.S., Markel, M.E., Garcia-Mazcorro, J.F., Unterer, S., Heilmann, R.M., Dowd, S.E., Kachroo, P., Ivanov, I., Minamoto, Y., Dillman, E.M., Steiner, J.M., Cook, A.K. & Toresson, L. (2012b). The Fecal Microbiome in Dogs with Acute Diarrhea and Idiopathic Inflammatory Bowel Disease. Plos One, vol. 7 (12), s. e51907.
Suchodolski, J.S., Xenoulis, P.G., Paddock, C.G., Steiner, J.M. & Jergens, A.E. (2010). Molecular analysis of the bacterial microbiota in duodenal biopsies from dogs with idiopathic inflammatory bowel disease. Veterinary Microbiology, vol. 142 (3–4), ss. 394–400.
15
https://en.wikipedia.org/w/index.php?title=Turicibacter&oldid=825612786. [Accessed 2019-03-03].
Van House, A.M., Palculict, B., Steiner, J.M., Xenoulis, P.G., Suchodolski, J.S. & Allenspach, K. (2008). Molecular-phylogenetic characterization of microbial
communities imbalances in the small intestine of dogs with inflammatory bowel disease.
FEMS Microbiology Ecology, vol. 66 (3), ss. 579–589. VetBact u.å.a Available from:
http://www.vetbact.org/index.php?artid=141&vbsearchstring=acinetobacter. [Accessed 2019a-03-01].
VetBact u.å.b Available from:
http://www.vetbact.org/index.php?artid=65&vbsearchstring=Pseudomonas%20aerugi nosa. [Accessed 2019b-03-01].
VetBact u.å.c Available from:
http://www.vetbact.org/index.php?artid=25&vbsearchstring=Clostridium. [Accessed 2019c-03-01].
VetBact u.å.d Available from: http://www.vetbact.org/index.php?artid=68. [Accessed
2019d03-01].
VetBact u.å.e Available from:
http://www.vetbact.org/index.php?artid=103&vbsearchstring=fusobacterium. [Accessed 2019e-03-03].
Xenoulis, P.G., Palculict, B., Allenspach, K., Steiner, J.M., Van House, A.M. & Suchodolski, J.S. (2008). Molecular-phylogenetic characterization of microbial communities imbalances in the small intestine of dogs with inflammatory bowel disease. FEMS
Microbiology Ecology, vol. 66 (3), ss. 579–589.