Examensarbete Biomedicinsk
Laboratorievetenskap Malmö högskola
15 hp Hälsa och samhälle
Biomedicinsk analytikerprogrammet 205 06 Malmö
Hälsa och samhälle
UTVECKLING AV EN PCR
METOD FÖR
IDENTIFIERING AV
NYUPPTÄCKTA
MJÖLKSYRABAKTERIER
MARIA CELANDER
UTVECKLING AV EN PCR
METOD FÖR
IDENTIFIERING AV
NYUPPTÄCKTA
MJÖLKSYRABAKTERIER
MARIA CELANDER
Celander, M. Utveckling av en PCR metod för identifiering av nyupptäckta mjölksyrabakterier. Examensarbetei Biomedicinsk Laboratorievetenskap 15 högskolepoäng. Malmö högskola: Hälsa och Samhälle, enheten för Biomedicinsk
laboratorievetenskap, 2011.
Flera olika arter av mjölksyrabakterier som ingår i släktena Lactobacillus och
Bifidobacterium har hittats hos bin och i deras honung. Idag finns ingen effektiv
metod för identifiering av bakterierna. Syftet med detta projekt är att utveckla en metod för snabb identifiering genom att hitta lämpliga primers till olika
mjölksyrabakterier och därmed få fram en Polymeraskedjereaktion (PCR) metod. Ribosomal ribonukleinsyra (rRNA) generna eller 16S-23S rRNA intergenic spacer region (ISR) används ofta vid design av primers, som därefter används i PCR för att identifiera olika bakterier. Deoxiribonukleinsyra (DNA) visualiseras i agarosgelen med hjälp av SYBRgreen I som fluorescens på ultraviolett (UV)-ljusbord. I detta projekt har 16S rRNA och 16S-23S rRNA ISR amplifierats i enkel PCR och multiplex PCR och visualiserats i agarosgel i försök att identifiera mjölksyrabakterierna. 16S rRNA har visat sig ha mycket liten variation mellan bakterierna och ansågs därför inte lämplig att använda för identifiering av närbesläktade arter. 16S-23S rRNA ISR visade större variation, fram för allt mellan lactobacillerna och bifidobakterierna. Gruppering av bakterierna med hjälp av multiplex PCR gjordes med viss framgång, med undantag av några bakterier som inte hamnade i den förväntade gruppen. Dock behövs fler försök för att stödja dessa resultat.
Nyckelord: Mjölksyrabakterier, polymeraskedjereaktion (PCR), primers, 16S ribosomal ribonukleinsyra (rRNA), 16S-23S rRNA intergenic spacer region (ISR).
DEVELOPMENT OF A PCR
METHOD FOR
IDENTIFICATION OF
NEWLY DISCOVERED
LACTIC ACID
BACTERIUM
MARIA CELANDER
Celander, M. Development of a PCR method for identification of newly
discovered lactic acid bacterium. DegreeProject, Biomedical Laboratory Science 15 Credit points. Malmö University: Health and Society, Department of
Biomedical Laboratory Science, 2011.
Several different lactic acid bacterium (LAB) species from the genera
Lactobacillus and Bifidobacterium was discovered in bees and in their honey.
Today there is no rapid and reliable method to identify these LAB. Therefore a rapid polymerase chain reaction (PCR) method to identify the LAB is needed. The aim of this project is to find primers suitable for the different LAB. Ribosomal ribonucleic acid (rRNA) genes or 16S-23S rRNA intergenic spacer region (ISR) are often used to designing of primers followed by PCR assays, for identification of different bacteria. To visualize deoxyribonucleic acid (DNA) in agarose gels, SYBRgreen I was used as fluorescence and then viewed under ultraviolet (UV) light. In this project the 16S rRNA and 16S-23S rRNA ISR was used as a target in a PCR and a multiplex PCR amplification. The PCR product was analyzed in agarose gel in an attempt to identify the LAB. 16S rRNA sequence have to little variation and is not suitable to identify closely related species. 16S-23S rRNA ISR sequence exhibits greater variations, especially between Lactobacillus and
Bifidobacterium. Differentiation of the bacteria into groups by multiplex PCR was
done with good result, except for some of the bacteria that did not end up in the expected group. More studys is needed to support these results.
Keywords: Lactic acid bacterium (LAB), polymerase chain reaction ( PCR), primers, 16S ribosomal ribonucleic acid (rRNA), 16S-23S rRNA intergenic spacer region (ISR).
INNEHÅLLSFÖRTECKNING
SAMMANFATTNING 1 ABSTRACT 2 INNEHÅLLSFÖRTECKNING 3 INLEDNING 4 Syfte 5MATERIAL OCH METOD 6
Material 6 Metod 7 RESULTAT 9 DISKUSSION 13 REFERENSER 15 BILAGA 16
INLEDNING
En forskargrupp i Lund, har upptäckt 13 nya mjölksyrabakterier varav 12 bakterier är publicerade, som ingår i släktena Lactobacillus och Bifidobacterium hos bin och i deras honung. Mjölksyrabakterier återfinns ofta hos friska individer och de används även i livsmedel och i probiotika [1,2]. Bakterierna anses vara nyttiga och de förmodas ha en viktig roll i regleringen av tarmens normalflora [1,3].
Vid identifiering av olika bakterier kan generna för ribosomal ribonukleinsyra (rRNA) användas. Vid tidigare studier har primers designats för gener tillhörande både 16S rRNA och 23S rRNA. Med polymeraskedjereaktion (PCR) har man därefter kunnat identifiera olika Lactobacillus arter [3].
16S rRNA genen innehåller 9 stycken hypervariabla regioner som skiljs åt av konserverade regioner (fig. 1). Det är de hypervariabla regionerna som ger variation mellan olika bakterier och som kan användas vid artidentifikation. De konserverade regionerna är emellertid målet vid design av primers för att därefter amplifiera någon eller några hypervariabla regioner [4]. Vid väldigt närbesläktade arter har 16S rRNA genen en liten variation och det är då svårt att skilja arterna åt med denna gen. Sekvensen från 16S-23S rRNA intergenic spacer region (ISR) kan ge större variation och är lämpligare mål för design av primers [3]. Sekvensen 16S-23S rRNA kan variera både i antal nukleotider och komposition av dessa, och kan därför vara lämplig vid design av art-specifika primers eller vid användning av multiplex PCR-metodik för identifiering av närbesläktade arter [5].
PCR-metod används för att amplifiera ett specifikt deoxiribonukleinsyra (DNA) fragment. Den består av tre steg, denaturering, annealing och elongering, där temperaturen skiljer i varje steg. Dessa steg repeteras i s k cykler. I annealing steget binder primers in och temperaturen vid detta steg bestäms vid varje PCR-metod. Temperaturen måste vara optimal, och om den inte är det, kan resultatet bli ospecifik inbindning eller avsaknad av DNA produkt. Det viktiga i
PCR-metodiken är design av de två oligonukleotid primerna; en forward och en reverse. Det krävs kännedom om sekvensen av fragmentet som ska amplifieras. Primerna designas utifrån sekvensen och dessa behöver vara komplementära till sekvensen, men ska inte vara komplementära till varandra. Dessutom måste de matcha varandra i antal guanin och cytosin (G+C) vilket styr
annealing-temperaturen [6].
Vid tidigare studier har primers designade för16S och 23S rRNA gener och 16S-23S rRNA ISR framgångsrikt använts vid multiplex PCR för identifiering av lactobacillusarter. Multiplex PCR har fördel av att vara en pålitlig, snabb och känslig analysmetod [7].
En vanlig metod för att separera DNA är elektrofores i agarosgel. Agaros används vanligen i koncentration mellan 1 % och 3 %. Porstorleken i gelen beror på koncentrationen av agaros. Låg koncentration agaros ger stor porstorlek. Olika koncentrationer i gelen används för olika stora DNA molekyler DNA är en negativt laddad molekyl och rör sig mot anoden i elektroforesen. Då de största molekylerna möter störst motstånd och de minsta molekylerna möter minst
motstånd i gelen så separeras DNA molekylerna efter storlek. Agaros har fördelen av låg smältpunkt, 62-65°C. DNA molekyler som separerats i gelen kan skäras ut
ur gelen, därefter kan gelerna smältas på nytt, genom att man värmer upp dem igen till 65°C och de blir åter flytande. På detta sätt kan man få fram en DNA sekvens som kan användas igen vid ny PCR eller vid sekvensering [6].
Att använda ethidium bromid (EtBr) för visualisering av DNA i gelen och därefter titta på den under ultraviolett ljus är en vanlig metod [6]. Ethidium bromid är både giftigt och mutagent och därför inte en idealisk metod. SYBRgreen I har visat hög affinitet för DNA och stor fluorescence vid UV-belysning. SYBRgreen I är ett färgämne som binder till dubbelsträngat DNA men har även förmåga att selektivt binda in till enbart dubbelsträngat DNA även i närvaro av ex RNA, enkelsträngat DNA och proteiner. Denna metod har visat sig fördelaktig vid låga DNA
koncentrationer [8].
Tidigare har de 13 mjölksyrabakteriernas 16S rRNA gen sekvenserats och därefter grupperats genom fylogenetisk analys. Lactobacillerna räknas som en grupp och bifidobakterierna en annan grupp. De olika fylotyperna har placerats i ett
fylogenetiskt träd, baserat på likheterna i 16S rRNA genen (fig. 2) [2].
Syfte
Identifiering av de 13 nyupptäckta mjölksyrabakterierna genom 16S rDNA PCR amplifiering och DNA sekvensering är en kostsam metod. Syftet med detta projekt är att utveckla en bättre metod genom att hitta lämpliga primers till de olika mjölksyrabakterierna och därmed få fram en enkel och snabb PCR samt en multiplex PCR för att identifiera bakterierna.
Figur 1. 16S rRNA´s sekundära struktur, där L. acidophilus används som modell [9]. De hypervariabla regionerna är märkta V1-V9 [10].
V1 V2 V3 V4 V5 V6 V7 V8 V9
Bin2 Bin7 Bifidobakterier Hma3 Bma6 Bma5 Hma8 Biut2 Hma2 Hma11 Lactobaciller Hon2 Bin4 Fhon2 Fhon13
Figur 2. Fylogenetiskt träd baserat på likheter i 16S rRNA genen. Efter tidigare studier [2].
MATERIAL OCH METOD
Material
13 mjölksyrabakterier av de olika fylotyperna; Bin7, Bin2, Bma6, Hma3, Hma2, Fhon2, Hma11, Fhon13, Biut2, Bma5, Bin4, Hon2, Hma8, odlade i MRS-buljong (MRS, fruktos och L-cysteine), eppendorfrör, glassand (106µm).
PCR
Reaktions buffert, nukleotid mix, taq polymeras (Roche, Mannheim, Germany) AmpliTaq Gold 360 buffert, magnesiumklorid, 360 GC enhancer, och AmpliTaq Gold 360 polymerase (Applied Biosystems, Foster city, USA)
PCR-rör, Thermo-fast 96, low profile 0,2ml (Thermo Scientific, Epsom, UK) Lock till PCR-rör (Thermo Scientific, Epsom, UK)
PCR (eppendorf mastercycler, Hamburg, Germany)
Agaroselektrofores
TB-buffert 10x (53,89g Tris base, 27,53g Borsyra, 4,66g EDTA, fylldes upp med sterilt vatten till 0,5 liter).
Loading buffert 6x (3ml glycerol, 25mg bromphenol blue, fylldes upp med sterilt vatten till 10ml).
DNA molekylvikts markör VI (Boehringer mannheim, Germany) 154, 220, 234, 298, 394, 453, 517, 653, 1033, 1230, 1766 och 2176 baspar (bp).
SYBRgreen I (Sigma-Aldrich, St. Louise, USA)
Agaros (Electran VWR international, Lutterworth, UK) Spänningsaggregat (Bio-Rad model 250/2.5)
Tabell 1. Primers som använts i projektet. Primers Sekvens (5´-3´) RP6 FP6 U4F ENV1 ENV2 16 23-10C Ldel-7 LU-1 LU-3 LU-5 Lac-2 ATTACCGCGGCTGCTGG 40-bp GC clamp-CCTACGGGAGGCAGCAG GTAGTCCACGCCGTAAACG AGAGTTTGATIITGGCTCAG* CGGITACCTTGTTACGACTT* GCTGGATCACCTCCTTTC CCTTTCCCTCACGGTACTG ACAGATGGATGGAGAGCAGA ATTGTAGAGCGACCGAGAAG AAACCGAGAACACCGCGTT AAACCGAGAACACCGCGTT CCTCTTCGCTCGCCGCTACT Ljen-3 AAGAAGGCACTGAGTACGGA Laci-1 TGCAAAGTGGTAGCGTAAGC Lcri-1 AGGATATGGAGAGCAGGAAT Lgas-1 AGCGACCGAGAAGAGAGAGA Lcri-2 CAACTATCTCTTACACTGCC Lgas-2 TGCTATCGCTTCAAGTGCTT * variabel. Metod
Fysisk sönderdelning av bakterierna
0,5ml av varje bakterieart odlade i MRS-buljong överfördes till 2ml eppendorfrör. Rören centrifugerades 13000rpm i 10 min (Biofuge pico). Därefter hälldes
supernatanten av och 0,5ml sterilt vatten blandades med pelleten. Detta upprepades två gånger och sista gången fylldes rören med 250µl.
4x13 stycken rör fylldes med ca 3mm glassand (106µm). Till varje rör tillsattes 100µl sterilt vatten samt 50µl bakteriesuspension. Rören placerades på en vortex anpassad för microtiterplattor på microtiter hastighet i 45 min (IKA, MS1
minishaker). Samtliga rör centrifugerades 2 min (VWR Galaxy mini) och placerades därefter i frys (-20°C).
V3 regionen
I ett autoklaverat rör tillsattes 32,75µl sterilt vatten, 5µl AmpliTaq Gold 360 buffert, 3µl 25Mm magnesiumklorid, 5µl 360 GC enhancer, 1µl av varje primer (RP6 och FP6), 1µl PCR nukleotid mix, 0,25µl AmpliTaq Gold 360 polymerase/ prov. Innehållet blandades med vortex. 49µl master mix pipetterades i PCR rör/prov och därefter tillsattes 1µl templat/prov, förutom ett rör utan DNA-templat som negativ kontroll vid elektrofores.
För att amplifiera V3 regionen i 16S rRNA genen användes ett PCR program som startade med ett prePCR steg 95°C 10 min, därefter följde denaturering 1 min 95°C, samt annealing och elongering 1 min på 56°C. Detta upprepades i 35 cykler och avslutades med en extra elongering på 72°C 10 min.
Standard
I ett autoklaverat rör tillsattes 40,5µl sterilt vatten, 5µl PCR reaktions buffert, 1µl av varje primer (reverse och forward), 1µl PCR nukleotid mix, 0,5µl Taq
i PCR rör/ prov och därefter tillsattes 1µl DNA-templat/prov, förutom ett rör utan DNA-templat som negativ kontroll vid elektrofores.
För att amplifiera hela 16S rRNA genen (primer ENV1 och ENV2) eller delar av 16S rRNA genen (primer ENV1 och RP6 eller U4F och ENV2) användes ett PCR program som innehöll denaturering 15 sek 96°C, annealing 30 sek 48°C och elongering 1 min och 30 sek 72°C. Detta upprepades i 30 cykler och avslutades med en extra elongering 72°C 10 min. För att rena upp PCR produkten
amplifierades hela 16S rRNA (primer ENV1 och ENV2) därefter gjordes en ny PCR på produkten med primer ENV1 och RP6. Samma program som ovan användes i båda fallen. För att amplifiera sekvensen 16S-23S rRNA ISR (primer 16 och 23-10C). användes ett PCR program som innehöll denaturering 20 sek 95°C, annealing och elongering 2 min 55°C. Detta upprepades i 35 cykler. Försök gjordes även med annealing och elongerings temperatur på 57°C och 58°C.
PCR-G
För att gruppera bakterierna användes standard master mix (se ovan) med
undantag av att 5 stycken primer blandades i en primermix bestående av följande primers: Ldel-7, LU-1, LU-3, LU-5 och Lac-2. 0,5µl av primer mixen
användes/prov. Denna master mix testades även med 1µl primer mix/ prov och 3µl primer mix/ prov. Vid 3µl primer mix ökades även DNA-templatet till 5µl/prov. Slutvolymen reglerades med vattenmängden till 50µl. I PCR
programmet som användes ingick denaturering 20 sek 95°C samt annealing och elongering 2 min 55°C. Detta upprepades i 35 cykler och avslutades med en extra elongering 74°C 5 min.
PCR-I och PCR-II
För artidentifiering användes standard master mix (se ovan) med undantag av att 3 respektive 4 stycken primer blandades i en primermix bestående av följande primers: Ljen-3, Laci-1 och 23-10C till PCR-I. Lcri-1, Lgas-1, Lcri-2 och Lgas-2 till PCR-II. 3µl av respektive primer mix användes/prov och 5µl DNA-templat/ prov, förutom ett rör utan DNA-templat som negativ kontroll vid elektrofores. Slutvolymen reglerades med vattenmängden till 50µl. I PCR programmen som användes ingick denaturering 20 sek 95°C samt annealing och elongering 2 min 68°C för PCR-I respektive 61°C för PCR-II. Detta upprepades i 35 cykler och avslutades med en extra elongering 74°C 5 min.
Gelelektrofores
Agarosgeler med 1-2% agaros tillverkades genom att lösa upp agaros i 1x TB-buffert.
10µl PCR produkt och 5µl SYBRgreen blandades i ett rör. 2µl sterilt vatten, 3µl molekylviktsstandard och 5µl SYBRgreen blandades också. Rören ställdes mörkt i 15 min så SYBRgreen kunde binda in till DNA. Till respektive brunn i gelen tillsattes 15µl prov, 3µl loading buffert samt 10µl molekylviktsstandard till separat brunn, dessutom tillsattes 15µl negativ kontroll till en brunn i gelen. PCR-produkt fick vandra i 1 tim och 45 min till 2 tim och 30 min vid en spänning av 100-120V. Därefter fotograferades gelerna på ett UV-ljusbord
RESULTAT
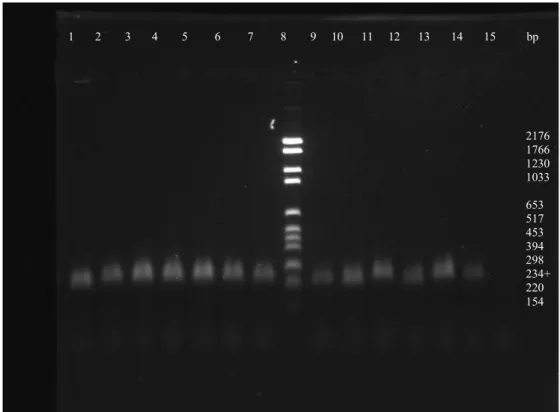
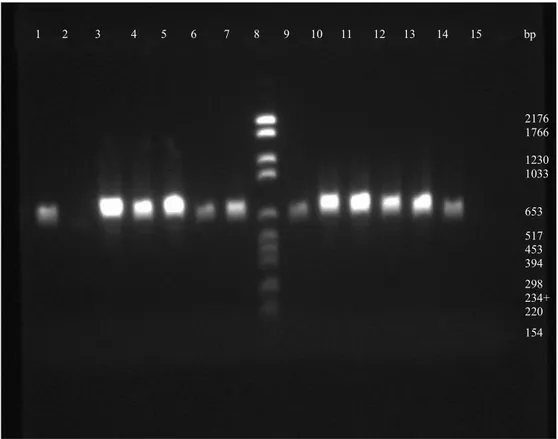
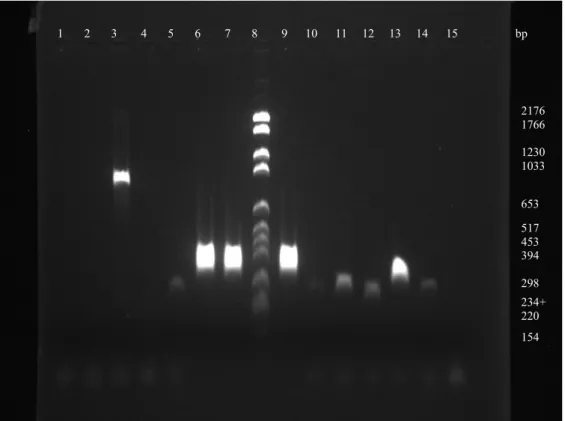
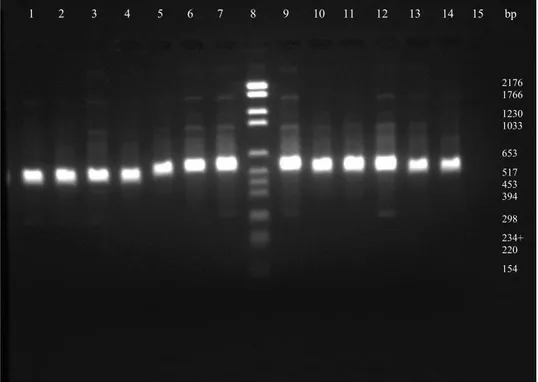
Fragmenten för V3 regionen visade mycket liten skillnad. Samtliga fragment låg på en storlek av ca 250bp (fig. 3). Fragmenten från V1, V2 och V3 var större ca 450-520bp, man kunde se skillnad på fragmentstorleken (fig. 4). När PCR produkten renades upp placerades bifidobakterierna ( Bin2, Bin7, Hma3 och Bma6 ) först vars fragment var något mindre än lactobacillernas fragment (fig. 5). V5, V6, V7, V8 och V9 regionen visade inte någon skillnad i fragmentstorlek. Fragmentstorleken låg på ca 650bp på samtliga bakterier (fig. 6). Sekvensen 16S-23S rRNA ISR visade stor skillnad mellan bifidobakterierna och lactobacillerna. Där fanns flera band som visade ospecifik inbindning av primerna (fig. 7). Gruppering av bakterierna med primermix PCR-G visade stor skillnad på fragmentstorlekarna. Fragment från 3 stycken bifidobakterier saknades, däremot hade bifidobakterien Bma6 ett fragment på ca 1000bp. Bma5, Hma8, Biut2, Hma2 och bin4 hade ett fragment på ca 250bp och Hon2 hade ett fragment på ca 300bp. Hma11, Fhon2 och Fhon13 hade samtliga ett fragment på ca 400bp (fig. 8).
Amplifieringen på artnivå saknade fragment på samtliga bakterier (fig. 9 och 10).
Figur 3. Amplifiering av V3 regionen. 1,5% agarosgel, 120V 1 tim och 45 min. I ordning från vänster till höger:1=Bin7, 2=Hma2, 3=Fhon2, 4=Hma11, 5=Fhon13, 6=Biut2, 7=Bma5, 8=standard, 9=Bin2, 10=Bma6, 11=Bin4, 12=Hma3, 13=Hon2, 14=Hma8 och
15= negativ kontroll. 2176 1766 1230 1033 653 517 453 394 298 234+ 220 154 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 bp
Figur 4. Amplifiering av V1, V2 och V3 regionen (primer ENV1 och RP6). 1,5% agarosgel, 120V 1 tim och 45 min. I ordning från vänster till höger: 1=Bin7, 2=Hma2, 3=Fhon2, 4=Hma11, 5=Fhon13, 6=Biut2, 7=Bma5, 8=standard, 9=Bin2, 10=Bma6, 11=Bin4, 12=Hma3, 13=Hon2, 14=Hma8 och 15=negativ kontroll.
Figur 5. Amplifiering av V1, V2 och V3 regionen (ENV1 och RP6). Från PCR produkten av hela 16S rRNA (ENV1 och ENV2). 1,5% agarosgel, 120V 1 tim och 45 min. I ordning från vänster till höger: 1=Bin7, 2=Bin2, 3=Bma6, 4=Hma3, 5=Hma2, 6=Fhon2, 7=Hma11, 8=standard, 9=Fhon13, 10=Biut2, 11=Bma5, 12=Bin4, 13=Hon2, 14=Hma8 och 15=negativ kontroll.
2176 1766 1230 1033 653 517 453 394 298 234+ 220 154 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 bp 2176 1766 1230 1033 653 517 453 394 298 234+ 220 154 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 bp
Figur 6. Amplifiering av V5, V6, V7, V8 och V9 regionen (primer U4F och ENV2). 1,5% agarosgel, 120V 1 tim och 45 min. I ordning från vänster till höger: 1=Bin7, 2=Hma2, 3=Fhon2 4=Hma11, 5=Fhon13, 6=Biut2, 7=Bma5, 8=standard, 9=Bin2, 10=Bma6, 11=Bin4, 12=Hma3, 13=Hon2, 14=Hma8 och 15=negativ kontroll.
Figur 7. Amplifiering av 16S-23S rRNA intergenic spacer region (ISR) (primer 16 och 23-10C). 1,5% agarosgel, 120V 1 tim och 45 min. I ordning från vänster till höger: 1=Bin7, 2=Bin2, 3=Bma6, 4=Hma3, 5=Hma2, 6=Fhon2, 7=Hma11, 8=standard, 9=Fhon13, 10=Biut2, 11=Bma5, 12=Bin4, 13=Hon2, 14=Hma8 och 15=negativ kontroll.
2176 1766 1230 1033 653 517 453 394 298 234+ 220 154 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 bp 2176 1766 1230 1033 653 517 453 394 298 234+ 220 154 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 bp
Figur 8. Gruppering av bakterierna (primer Ldel-7, LU-1, LU-3, LU-5 och Lac-2). 1,5 % agarosgel, 120V 1 tim och 45 min. I ordning från vänster till höger: 1=Bin7, 2=Bin2, 3=Bma6, 4=Hma3, 5=Hma2, 6=Fhon2, 7=Hma11, 8=standard, 9=Fhon13, 10=Biut2, 11=Bma5, 12=Bin4, 13=Hon2, 14=Hma8 och 15=negativ kontroll.
Figur 9. Amplifiering på artnivå (primer Ljen-3, Laci-1och 23-10C). 1,5 % agarosgel, 120V 1 tim och 45 min. I ordning från vänster till höger: 1=Bin7, 2=Bin2, 3=Bma6, 4=Hma3, 5=Hma2, 6=Fhon2, 7=Hma11, 8=standard, 9=Fhon13, 10=Biut2, 11=Bma5, 12=Bin4, 13=Hon2, 14=Hma8 och 15=negativ kontroll.
2176 1766 1230 1033 653 517 453 394 298 234+ 220 154 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 bp 2176 1766 1230 1033 653 517 453 394 298 234+ 220 154 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 bp
Figur 10. Amplifiering på artnivå (primer Lcri-1, Lgas-1, Lcri-2 och Lgas-2). 1,5 % agarosgel, 120V i 1 tim och 45 min. I ordning från vänster till höger: 1=Bin7, 2=Bin2, 3=Bma6, 4=Hma3, 5=Hma2, 6=Fhon2, 7=Hma11, 8=standard, 9=Fhon13, 10=Biut2, 11=Bma5, 12=Bin4, 13=Hon2, 14=Hma8 och 15=negativ kontroll.
DISKUSSION
V3 regionen visade ingen eller mycket liten variation hos samtliga bakterier (fig. 3). Inte heller V5-V9 regionen visade någon större skillnad i antal bp mellan bakterierna (fig. 6). Däremot kunde man se skillnad i antal bp mellan lactobaciller och bifidobakterier vid amplifiering av V1-V3 regionen (fig. 4 och 5). Detta visade att den största skillnaden i 16S rRNA mellan de 13 bakterierna finns i V1-V3 regionen. Det visade också att 16S rRNA inte är lämplig vid identifiering av bakterierna på artnivå utan endast på släktnivå. Vid amplifieringen av V1-V3 regionen från PCR produkten från hela 16S rRNA kunde man se svaga band som kunde bero på ospecifik inbindning. Det kunde även bero på en kontamination av främmande DNA orsakad vid blandning av mastermix (fig. 5).
16S-23S rRNA ISR visade stora skillnader. Fram för allt mellan lactobaciller och bifidobakterier. Förutom de tjocka banden hos lactobacillerna, så fanns det flera andra tunna band. Detta tyder på ospecifik inbindning. Även bifidobakterierna visade på ospecifik inbindning, men till skillnad från lactobacillerna så saknade bifidobakterierna ett tjockt och markant band (fig. 7). Vid tidigare studier på lactobacillarter har man amplifierat 16S-23S rRNA ISR för att använda sekvensen för design av primers [3]. Det skulle vara lämpligt även vid detta projekt. På så sätt skulle man få möjlighet att designa primers direkt riktade till de olika arterna.
2176 1766 1230 1033 653 517 453 394 298 234+ 220 154 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 bp
Där man först grupperade lactobacillerna i en första PCR och artidentifierade i en andra PCR. Vilken grupp lactobacillerna tillhörde, avgjorde vilka primers som skulle användas i en andra PCR [3]. Även i detta projekt grupperades
mjölksyrabakterierna (fig 8).
Vid grupperingen av mjölksyrabakterierna saknades DNA från 3 stycken
bifidobakterier. Dessa primers hade inte använts tidigare för bifidobakterier utan endast för gruppering av lactobacillarter [3]. Bifidobakterier (Bin2, Bin7 och Hma3) är nära besläktade med varandra. Medan den fjärde bifidobakterien vars fragment låg på 1000bp (Bma6) inte är lika nära besläktad med de övriga
bifidobakterierna. Bma5, Hma8, Biut2, Hma2 och Bin4 hade en fragmentstorlek på 250bp. De fyra förstnämnda är nära besläktade medan den femte (Bin4) kanske borde haft en fragmentstorlek på 300bp, då den är närmre släkt med Hon2 som hade en fragmentstorlek på 300bp. Hma11, Fhon2 och Fhon13 hade alla en fragmentstorlek på 400bp. Fhon2 och Fhon13 är nära besläktade med varandra, medan Hma11 är närmre besläktade med Bma5, Hma8, Biut2 och Hma2 (fig 2 och 8). Detta försök borde upprepas då det finns risk att bakterierna har varit kontaminerade och då gett falska resultat. Man kunde även undersöka sekvenserna och på det sätt designa lämpligare primers.
Försöket med att artidentifiera bakterierna resulterade i inga fragment kunde detekteras. Detta kunde bero på att DNA inte renades upp utan bakterierna endast sönderdelades och i och med detta fanns en del nedbrytande enzymer kvar
(DNaser) som brutit ned DNA. Det kunde även bero på att primerna inte var optimala och därmed inte bundit in till DNA (fig 9 och 10).
Då visualisering av agarosgelerna gav ett utdraget utseende så gjordes försök med olika koncentrationer agaros i gelerna, utan någon förbättring. Även lägre
spänning under elektroforesen under längre tid testades. Inte heller detta gav ett bättre resultat (se bilaga, fig. 1-3).
P g a ospecifik inbindning av primerna vid amplifiering av 16S-23S rRNA ISR (fig 7), testades olika annealing temperaturer. Förutom 55°C, gjordes försök med 57°C och 58°C. Dessa två sistnämnda försök gjordes endast på 6 stycken
lactobaciller och 2 stycken bifidobakterier. Eftersom det inte hade någon annan avsikt än att se om primerna band in mer specifikt. Ingen förbättring kunde ses (se bilaga, fig 4-5).
REFERENSER
1. Vásquez A, Olofsson T C (2009) The lactic acid bacteria involved in production of bee pollen and bee bread. Journal of Apicultural Research
and Bee World, 48(3); 189-195
2. Olofsson T C, Vásquez (2008) Detection and Identification of a Novel Lactic Acid Bacterial Flora Within the Honey Stomach of the Honeybee
Apis mellifera. Current Microbiology, 57; 356-363
3. Song Y-L, Kato N, Liu C-X, Matsumiya Y, Kato H, Watanabe K (2000) Rapid identification of 11 human intestinal Lactobacillus species by multiplex PCR assays using group- and species-specific primers derived from the 16S-23S rRNA intergenic spacer region and its flanking 23S rRNA. FEMS Microbiology Letters, 187; 167-173
4. Chakravorty S, Helb D, Burday M, Connell N, Alland D (2007) A detailed analysis of 16S ribosomal RNA gene segments for the diagnosis of
pathogenic bacteria. Journal of Microbiological Methods, 69; 330-339 5. Ferchichi M, Valcheva R, Prévost H, Onno B, Dousset X (2008) A
one-step reaction for rapid identification of Lactobacillus mindensis,
Lactobacillus panis, Lactobacillus paralimentarius, Lactobacillus pontis
and Lactobacillus frumenti using oligonucleotide primers designed from the 16S-23S rRNA intergenic sequences. Journal of Applied
Microbiology, 104(6); 1797-1807
6. Wilson K, Walker J (2000) Principles and Techniques of Practical
Biochemistry, Cambridge: Cambridge university press.
7. Nikolaou A, Saxami G, Kourkoutas Y, Galanis A (2011) A new methodology for rapid detection of Lactobacillus delbrueckii subsp.
bulgaricus based on multiplex PCR. Journal of Microbiological Methods, 84; 362-364
8. Rengarajan K, Cristol S M, Mehta M, Nickerson J M (2002) Quantifying DNA concentrations using florometry: A comparison of fluorophores.
Molecular Vision, 8; 416-421
9. The University of Texas at Austin. Comparative RNA web site and project/ secondary structure diagram Retrieval.
http://www.rna.ccbb.utexas.edu/DAT/3C/Structure/index.php
hämtades: 2011-04-21
10. Neefs JM, Van de Peer Y, De Rijk P, Chapelle S, De Wachter R (1993) Compilation of small ribosomal subunit RNA structures. Nucleic Acids
BILAGA
Figur 1. Amplifiering av V1, V2 och V3 regionen (primer ENV1 och RP6). 1,0 % agarosgel, 120V i 2 tim och 30 min. I ordning från vänster till höger: 1=Bin7, 2=Hma2, 3=Fhon2, 4=Hma11, 5=Fhon13, 6=Biut2, 7=Bma5, 8=standard, 9=Bin2, 10=Bma6, 11=Bin4, 12=Hma3, 13=Hon2, 14=Hma8 och 15=negativ kontroll.
Figur 2. Amplifiering av V3 regionen. 1,2 % agarosgel, 100V i 2 tim och 30 min. I ordning från vänster till höger: 1=Bin7, 2=Hma2, 3=Fhon2, 4=Hma11, 5=Fhon13, 6=Biut2, 7=Bma5, 8=standard, 9=Bin2, 10=Bma6, 11=Bin4, 12=Hma3, 13=Hon2, 14=Hma8 och 15=negativ kontroll.
2176 1766 1230 1033 653 517 453 394 298 234+ 220 154 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 bp 2176 1766 1230 1033 653 517 453 394 298 234+ 220 154 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 bp
Figur 3. Amplifiering av V1, V2 och V3 regionen (ENV1 och RP6). Från PCR produkten av hela 16S rRNA (ENV1 och ENV2). 2,0 % agarosgel, 120V i 1 tim och 45 min. I ordning från vänster till höger: 1=Bin7, 2=Bin2, 3=Bma6, 4=Hma3, 5=Hma2, 6=Fhon2, 7=Hma11, 8=standard, 9=Fhon13, 10=Biut2, 11=Bma5, 12=Bin4, 13=Hon2, 14=Hma8 och 15=negativ kontroll.
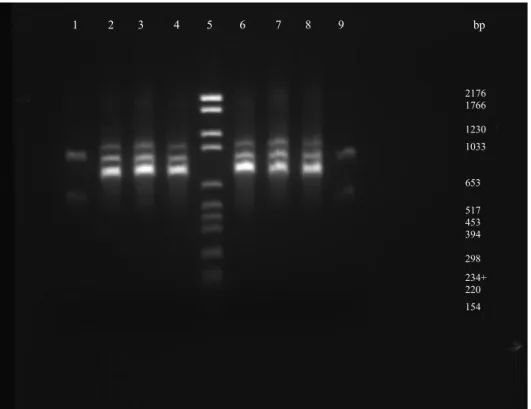
Figur 4. Agarosgel med PCR produkt från annealing temperatur på 57°C. 1,5 % agarosgel, 120V i 1 tim och 45 min. I ordning från vänster till höger: 1=Bin7, 2=Hma2, 3=Fhon2, 4=Hma11, 5=standard, 6=Fhon13, 7=Biut2, 8=Bma5, 9=Bin2.
2176 1766 1230 1033 653 517 453 394 298 234+ 220 154 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 bp 2176 1766 1230 1033 653 517 453 394 298 234+ 220 154 1 2 3 4 5 6 7 8 9 bp
Figur 5. Agarosgel med PCR produkt från annealing temperatur på 58°C. 1,5 % agarosgel, 120V i 1 tim och 45 min. I ordning från vänster till höger: 1=Bin7, 2=Hma2, 3=Fhon2, 4=Hma11, 5=standard, 6=Fhon13, 7=Biut2, 8=Bma5, 9=Bin2.
2176 1766 1230 1033 653 517 453 394 298 234+ 220 154 1 2 3 4 5 6 7 8 9 bp
![Figur 1. 16S rRNA´s sekundära struktur, där L. acidophilus används som modell [9]. De hypervariabla regionerna är märkta V1-V9 [10].](https://thumb-eu.123doks.com/thumbv2/5dokorg/3973262.77975/6.892.250.597.635.979/figur-sekundära-struktur-acidophilus-används-hypervariabla-regionerna-märkta.webp)
![Figur 2. Fylogenetiskt träd baserat på likheter i 16S rRNA genen. Efter tidigare studier [2]](https://thumb-eu.123doks.com/thumbv2/5dokorg/3973262.77975/7.892.312.730.85.363/figur-fylogenetiskt-träd-baserat-likheter-genen-tidigare-studier.webp)