Examensarbete i biomedicinsk laboratorievetenskap Malmö universitet
61-90 hp Hälsa och samhälle
Biomedicinska analytikerprogrammet 205 06 Malmö
September 2020
DETEKTION AV
CIPROFLOXACIN-RESISTENS
HOS
NEISSERIA GONORRHOEAE
MED PCR
DETEKTION AV
CIPROFLOXACIN-RESISTENS
HOS
NEISSERIA GONORRHOEAE
MED PCR
GABRIEL JENSEN ALAS
ABSTRAKT
Neisseria gonorrhoeae (NG) har successivt utvecklat resistens mot många
antimikrobiella medel och betraktas som ett av de tre reella hoten bland antibiotikaresistenta bakterier. Ciprofloxacin är ett bredspektrum-antibiotikum
tillhörande gruppen kinoloner som, förutom att behandla urinvägsinfektioner, används mot NG och infektioner i mage och tarm. Dock har det rapporterats att ca 30 % av NG-isolat som samlats in genom gonokock-isolatövervakningsprojekt (GISP) under 2017 var resistenta mot ciprofloxacin. På molekylnivå är resistens mot ciprofloxacin starkt associerad med en enda mutation i kodon 91 i gyras-genen (gyrA).
Detta projekt har undersökt om det går att använda molekylärbiologiska metoder för att detektera NG-isolat med gyrA mutationen. Analysen gjordes med två olika PCR-system, ”7500 Fast Real-Time PCR System” från Applied Biosystems (ABI) och Panther Fusion från Hologic. Proberna som användes designades för påvisning av vildtyp gyrA (ciprofloxacin-känslig) och mutant gyrA (ciprofloxacin-resistent) hos NG.
I projektet analyserades 50 NG-positiva prov (analyserade med screeningtest APTIMA COMBO2 från Hologic), från 43 patienter som provtagits under januari-februari 2020 i Region Skåne. Några patienter testades flera gånger vid olika tillfällen. NG-odling hade utförts parallellt från motsvarande tagna prov från patienterna.
ABI-metoden påvisade genen hos 90 % (45/50) av NG-positiva prover (APTIMA COMBO2) medan endast 24 av de 49 proven (49 %) kunde odlas med traditionell metodik för att därefter resistensbestämmas. Av de 45 prov där gyras-genen kunde detekteras med ABI-metoden, uppvisade 28 (62 %) av proven en muterad gen och därmed en potentiell resistens för ciprofloxacin.
Panther Fusion-metoden påvisade genen hos 80 % (40/50) av NG-positiva prover (APTIMA COMBO2), och såsom tidigare nämnts, kunde endast 24 av de 49 proven (49 %) odlas med traditionell metodik för att därefter resistensbestämmas. Av de 40 prov där gyras-genen kunde detekteras med Panther Fusion-metoden, uppvisade 26 av proven (65 %) en muterad gen och därmed en potentiell resistens för ciprofloxacin. De båda PCR-metoderna och traditionell odling uppvisade jämförbara resultat. Av de 24 prov som kunde odlas och därmed resistensbestämmas, detekterades med ABI-metoden gyras-genen i 23 av dessa prov och i 22 av proven med Panther
Fusion-metoden. Resistens mot ciprofloxacin uppvisades genom odling i 16 av de 24 odlingsbara prov, och av dessa 24 odlingsbara prov uppvisade ABI-metoden en muterad gen i 15 av proven och Panther Fusion-metoden en muterad gen i 14 av proven.
Traditionell odling kunde bara genomföras på 24 av proven och PCR-metoderna identifierade signifikant fler prov innehållande vildtyp eller muterad gyras-gen, 45 respektive 40 prov. Projektet visade tydligt att PCR-metoderna kan identifiera fler prov än genom traditionell odling och kan därmed upptäcka fler prov med förväntad ciprofloxacin-resistens än vad som kan bestämmas genom traditionell odling.
DETECTION OF
CIPROFLOXACIN-RESISTANCE
IN
NEISSERIA GONORRHOEAE
USING PCR
GABRIEL JENSEN ALAS
ABSTRACT
Neisseria gonorrhoeae (NG) has been developing a resistance towards several
different antibiotics and is viewed as one of the three real threats among resistant bacteria. Ciprofloxacin is a broad-spectrum-antibiotic belonging to the group quinolone antibiotics which, in addition to being used to treat urinal infections, is used to treat NG and infections in the stomach and intestines. However, it has been reported that 30 % of NG-isolates that have been gathered through the Gonococcal Isolate Surveillance Project (GISP) throughout 2017 were resistant to ciprofloxacin. On a molecular level, resistance to ciprofloxacin is strongly
associated with a single mutation in kodon 91 in the gyras-gene (gyrA). This project sought to examine if it is possible to use methods from molecular biology to detect which NG that have the gyrA-mutation. The test was done using two different PCR-systems, ”7500 Fast Real-Time PCR System” from Applied Biosystems (ABI) and Panther Fusion from Hologic. The probes used were designed to show wild type gyrA (ciprofloxacin sensitive), and mutated gyrA (ciprofloxacin resistant) in NG.
In this project 50 NG-positive samples (analysed with screentest APTIMA COMBO2 from Hologic), from 43 patients that had been tested during January-February 2020 in Region Skåne, were analysed. Some patients were tested several times, within the time period. NG-cultivation had been done in parallel from corresponding samples taken from the patients.
The ABI-method showed the gene in 90 % (45/50) of NG-positive samples (APTIMA COMBO2) while only 24 of the 49 samples (49 %) could be cultivated by traditional methodology, and then tested for resistance. Of the 45 samples where the gyras-gene could be detected with the ABI-method, 28 samples (62 %) exhibited a mutated gene and thus a potential resistance to ciprofloxacin.
The Panther fusion-method showed the gene in 80 % (40/50) of NG-positive samples (APTIMA COMBO2), and as mentioned earlier, only 24 of the 49 samples (49 %) could be cultivated by traditional methodology to then be tested for resistance. Of the 40 samples where the gyras-gene could be detected with the Panther Fusion-method, 26 samples (65 %) exhibited a mutated gene and thus a potential resistance to ciprofloxacin.
The two PCR-methods and traditional cultivation exhibited comparable results. Of the 24 samples that could be cultivated and thus tested for resistance, the ABI-method detected the gyras-gene in 23 of these samples and the Panther Fusion-method detected the gene in 22 of the samples. Cultivation exhibited resistance to ciprofloxacin in 16 of the 24 samples that could be cultivated, and of these 24 cultivatable samples the ABI method exhibited a mutated gene in 15 of the samples and the Panther Fusion-method exhibited a mutated gene in 14 of the samples.
Traditional cultivation could only be done on 24 of the samples and the PCR-methods could identify significantly more samples containing either wild type or mutated gyras-gene, 45 and 40 samples, respectively. The project clearly showed that more samples can be identified with the PCR-methods than through
traditional cultivation, and thereby discover more samples with expected
ciprofloxacin-resistance, than can be determined through traditional cultivation.
Innehållsförteckning
Bakgrund ... 1
Neisseria gonorrhoeae ... 1
Ciprofloxacin ... 1
Syfte ... 1
Material och metod ... 2
Patientmaterial ... 2
Positiva kontroller ... 2
Specificitetstest med DNA från NG-stammar ... 2
DNA extraktion till ABI ... 2
MgCl2 optimering Panther Fusion. ... 2
Prov för känslighets-test ... 2 Realtids PCR ABI ... 2 Panther fusion ... 3 Material ... 3 ABI ... 4 Panther fusion ... 5 Statistik ... 5 Resultat ... 6 MgCl2 optimering ... 6 Testning av prober ... 6
Känslighetsanalys Panther Fusion ... 7
Patientprov ... 8 ABI ... 8 Panther Fusion ... 8 Odling ... 10 Statistik ... 11 Diskussion ... 11 Konklusion ... 12 Referenser ... 13 Bilaga ... 14
1
BAKGRUND
Neisseria gonorrhoeae (NG) har successivt utvecklat resistens mot många
antimikrobiella medel och betraktas som ett av de tre reella hoten bland antibiotikaresistenta bakterier. Ciprofloxacin är ett bredspektrum-antibiotikum tillhörande gruppen kinoloner som, förutom att behandla urinvägsinfektioner, används mot NG och infektioner i mage och tarm [1]. Dock har det rapporterats att ca 30% av NG-isolat som samlats in genom gonokockisolatövervaknings-projekt (GISP) under 2017 var resistenta mot ciprofloxacin [2]. Förutom
traditionell odling för resistensbestämning kan resistens mot ciprofloxacin påvisas genom nukleinsyraamplifieringstest (NAAT) -baserad detektion direkt från kliniska prover [3]. På molekylnivå är resistens mot ciprofloxacin starkt
associerad med en enda mutation i kodon 91 i gyras-genen (gyrA) och mutationen har visat sig ha stark koppling till ciprofloxacin-resistens [3, 4]. Även om andra kodoner i gyrA, såväl som i topoisomeras IV (parC), och i sällsynta fall har penicillinbindande protein 2 (PBP2/ PenA) associerats med resistens mot ciprofloxacin, har majoriteten av ciprofloxacin-resistenta NG endast
GyrA-mutationen i kodon 91, vilket gör genen till ett mål för molekylär screening [3, 5]. Neisseria gonorrhoeae
En bakterie som trots effektiva antibiotikabehandlingar fortfarande är en vanlig orsak för infektion. Den är nummer två av alla sexuellt överförbara infektioner i världen. Bakterien fäster sig vid slemhinneceller och stimulerar produktionen av TNF-alfa, en proinflammation cytokin, som leder till de flesta av symptomen som kopplas till gonokocksjukdomar. [1]
Ciprofloxacin
Ett bredspektrumantibiotikum som tillhör gruppen kinoloner. Används vid urinvägsinfektion, infektioner i tarm och mage samt mot gonorré. Har en hämmande effekt på DNA-gyras, som är ett enzym som packar ihop och kan öppna den prokaryota kromosomen. [2]
Syfte
Syftet med projektet var att undersöka om NG-resistens mot ciprofloxacin kan påvisas med två olika molekylärbiologiska analysinstrument, Panther Fusion och ABI.
2
MATERIAL OCH METOD
Patientmaterial
Projektet analyserade 50 NG-positiva prov (analyserade med screeningtestet APTIMA COMBO2 från Hologic), från 43 patienter, några testades flera gånger vid olika tillfällen, som provtagits under januari-februari 2020 i Region Skåne. Proverna hade analyserats vid Klinisk Mikrobiologi Lund, och sedan sparats i frys. NG-odling hade utförts parallellt från motsvarande tagna prov från patienterna. Odling utfördes vid Klinisk Mikrobiologi Lund.
Positiva kontroller
För att säkerställa positiv PCR-signal från ciprofloxacin-känslig NG (även kallad viltyp-NG) och från ciprofloxacin-resistent NG användes positiva kontroller från odlade NG stammar (CCUG 15821, CCUG 57601 och CCUG 57602 för vildtyp NG, och CCUG 57597, GC 106 Res och GC 107 Res för resistent NG), där ca 10 kolonier från respektive stam slammades i provrör (Multitest Swab Transport Media, STM, Hologic). DNA från beredningarna extraherades enligt nedan.
Specificitetstest med DNA från NG-stammar
För att analysera att ciprofloxacin-resistent NG och känslig NG endast påvisas med respektive PCR för ciprofloxacin-resistens och för ciprofloxacin-känslighet användes NG stammarna ovan ((CCUG 15821, CCUG 57601 och CCUG 57602 för vildtyp NG, och CCUG 57597, GC 106 Res och GC 107 Res för resistent NG). Korsreaktivitet hos PCR testades även mot en Neisseria meningitidis-stam (ATCC 13090), där ca 10 kolonier slammades i provrör (Multitest Swab
Transport Media, STM, Hologic).
DNA extraktion till ABI
400 µL av prov och kontroller överfördes till 1,5 mL spetsiga Eppendorfrör (Sarstedt, Tyskland), och lämnades till personal som rutinmässigt utförde DNA-extraktion.
Extraktionen gjordes med extraktionsroboten Mag-Lead (Bioservice, Sverige) med program MagDEA Dx SV400 och 50 uL elueringsvätska genererades.
MgCl2 optimering Panther Fusion.
Open Access applikationen (Hologic) anger att optimal MgCl2 koncentrationska bestämmas för PCR som utförs på Panther Fusion instrumentet och tre olika PPR-mixar med slutkoncentrationerna 2 mM, 3 mM respektive 4 mM MgCl2 testades. De olika MgCl2 koncentrationerna analyserades med prover som innehöll
ciprofloxacin-känslig (20KO505189) respektive ciprofloxacin-resistent (20KO504956) NG.
Prov för känslighets-test
Spädningsserier med spädningsfaktor 1:10 gjordes med positiva kontrollerna CCUG 57601 och GC 106 Res. För att få en spädningsfaktor 1:10 överfördes 322 µL från originalröret till ett nytt Aptima-rör som redan innehöll 2,9 mL lösning. Nästa steg tog 322 µL från det nya röret till nästa Aptima-rör. Detta repeterades till 6 spädningar gjorts.
Realtids PCR ABI
ABI-metod använde 7500 Fast Real-Time PCR System från Applied Biosystems (ABI). Instrumentet är gjord för att kunna analysera många prover samtidigt med
3
hög sensitivitet [6]. Proberna (MGB) designades med hjälp av mjukvara PCR express (ABI, Version 3.0.1) med inställning TagMan®MGB Allelic
Discrimination.
Proberna beställdes från ABI och Eurofins.
Panther fusion
Instrumentet som användes var Panther Fusion från Hologic, vilken utför både nukleinsyraextraktion och Realtids-PCR [7]. Open Access är ett program från Hologic som kan användas på Panther Fusion för att analysera tester parallellt med diagnostiska kit-tester [8].
Metod som användes följde inställningar på Panther Fusion instrument som används vid Klinisk mikrobiologi. Förbrukningsmaterial som användes för analys tillhandahölls av handledare.
Material
Följande material och instrument användes till ABI metoden:
7500 Fast Real-Time PCR System från ABI (Foster City, USA), PCR Platta från ABI, Plast-film till PCR Platta från ABI, Taqman Universal PCR Master Mix från ABI. MagLead DNA extraktor från Bioservice (Munich, Tyskland) användes till att utföra DNA-extraktioner från prov före PCR med ABI. DNA extraktioner pipetterades till PCR-platta med Corbett robot från Qiagen (Hilden, Tyskland), Automat pipetter, Plattcentrifug Miniplate centrifuge (Aosheng).
Följande material och instrument användes till Panther Fusion metoden: Panther Fusion från Hologic (Marlborough, USA), Open Access program från Hologic, PCR Cartridge från Hologic, Swab Specimen Transfer provrör från Hologic, KCL 1000 mM från Hologic, MgCl2 1000 mM från Hologic, Tris HCl 1000 mM från Hologic, Intern Kontroll (IK) primers från Hologic, Intern Kontroll (IK) prob från Hologic, Automat pipetter, Mikrocentrifug Ministar från VWR (Radnor, USA).
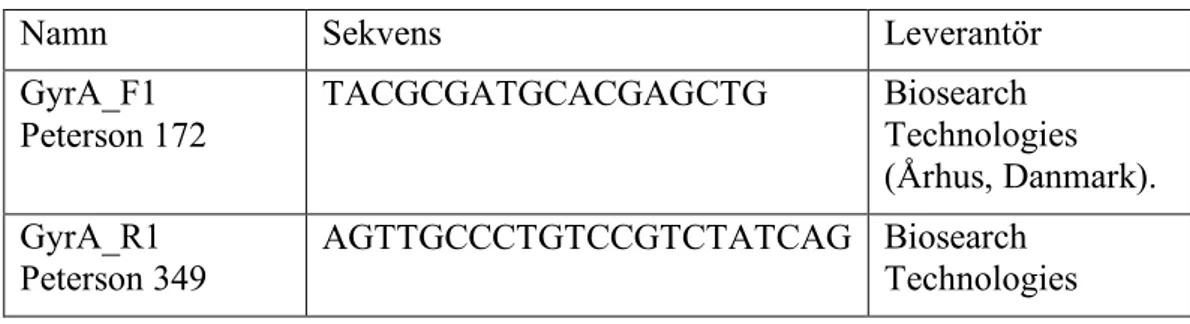
Primers som användes var samma som tidigare beskrivits av Peterson et al [9]. Primers namn, sekvens och leverantör visas i tabell 1.
Tabell 1. Namn, sekvens och leverantör för primers som användes för projektet.
Namn Sekvens Leverantör
GyrA_F1 Peterson 172 TACGCGATGCACGAGCTG Biosearch Technologies (Århus, Danmark). GyrA_R1
Peterson 349 AGTTGCCCTGTCCGTCTATCAG Biosearch Technologies
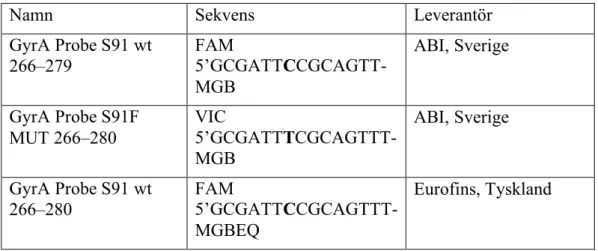
Proberna som används designades för påvisning av vildtyp och mutant NG-gyrA. Proberna designades med programmet Primer Express 3.0 som är gjort av ABI. Probernas namn, sekvens och leverantör visas i tabell 2.
4
Tabell 2. Namn, sekvens och leverantör för prober som användes för projektet.
Namn Sekvens Leverantör
GyrA Probe S91 wt 266–279 FAM 5’GCGATTCCGCAGTT-MGB ABI, Sverige GyrA Probe S91F MUT 266–280 VIC 5’GCGATTTCGCAGTTT-MGB ABI, Sverige GyrA Probe S91 wt 266–280 FAM 5’GCGATTCCGCAGTTT-MGBEQ Eurofins, Tyskland ABI
Först analyserades odlade NG-stammar, varav 3 ciprofloxacin-känsliga vildtyper (WT): CCUG 15821, CCUG 57601 och CCUG 57602, 3 ciprofloxacin-resistenta (MUT): CCUG 57597, GC 106 Res och GC 107 Res, samt 1 stam Neisseria
meningitidis: ATCC 13090. DNA från dessa prover extraherades med MagLead.
DNA extraktet pipetterades sedan med Corbett robot (Qiagen) till en PCR-platta med 96 brunnar som gjorts av ABI för Realtids-PCR i deras instrument ”7500 Fast Real-Time PCR System”. Varje brunn med prov innehöll 25 µl: 20 µl från PCR-mix och 5 µl från prov. Koncentrationerna för reagens till PCR-mixen som användes till ABI-metoden visas i tabell 3.
Tabell 3. Koncentration av primer och prober i PCR-mixen.
Reagens Work konc Konc i PCR µl i PCR
GyrA_F1 Peterson 172 10 µM 0,2 µM 0,5
GyrA_R1 Peterson 349 10 µM 0,2 µM 0,5
GyrA Probe S91 wt 266–280: FAM 6,25 µM 0,05 µM 0,2 GyrA Probe S91F MUT 266–280:
VIC
10 µM 0,05 µM 0,125
Taqman Universal PCR Master Mix 1 X 12,5
Vatten 6,175
Prov 5
Total mängd 25
Först beräknades volym av varje reagens till PCR-mixen genom att multiplicera mängderna i kolumnen ”µl i PCR” med antalet prover som skulle analyseras. Därefter blandades PCR-mix, från vilket det pipetteradess 20 µL av PCR-mixen till brunnar i PCR-platta. Efteråt pipetterades 5 µl av respektive prov till plattan. Efter att pipetteringen var klar täcktes den med en plast-film för PCR-plattor och centrifugerades i 30 sek med 2500 rpm (Miniplate centrifuge,Aosheng Labteamet, Helsingborg). PCR utfördes med instrumentet 7500 Fast Real-Time PCR System från ABI som analyserades i Standard mode med följande protokoll: uppvärmning på 95˚C i 15 min följt av 45 cykler av 95˚C i 15 sek och 60˚C i 60 sek.
5
Panther fusion
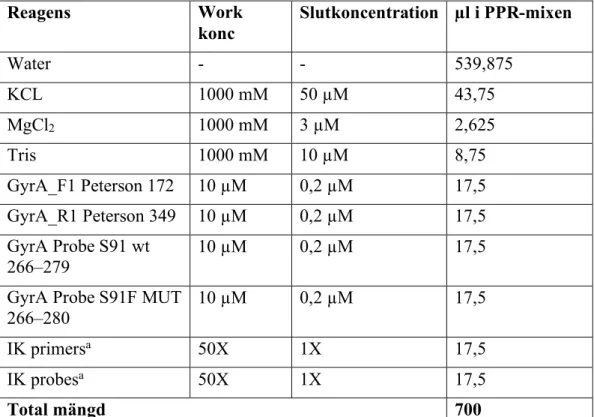
PCR-mixen som användes i instrumentet, kallad PPR-mix, hade koncentrationerna enligt tabell 4.
Tabell 4. Koncentrationerna för PPR-mixen som används i Panther fusion och mängderna för 18
analyser.
Reagens Work
konc Slutkoncentration µl i PPR-mixen
Water - - 539,875 KCL 1000 mM 50 µM 43,75 MgCl2 1000 mM 3 µM 2,625 Tris 1000 mM 10 µM 8,75 GyrA_F1 Peterson 172 10 µM 0,2 µM 17,5 GyrA_R1 Peterson 349 10 µM 0,2 µM 17,5 GyrA Probe S91 wt 266–279 10 µM 0,2 µM 17,5
GyrA Probe S91F MUT
266–280 10 µM 0,2 µM 17,5
IK primersa 50X 1X 17,5
IK probesa 50X 1X 17,5
Total mängd 700
a) Medföljande primers och probes i reagens (Hologic) för att kontrollera att PCR fungerat.
Efter att alla reagenser blandats i röret skakades röret följt av 5 sekunder centrifugering (6000 rpm med centrifug Ministar, VWR, Lund) som följdes av pipettering av 300 µl olja ovanpå mixen och ytterligare en snabb centrifugering. Denna mängd mix och olja var för 18 analyser. Resultat analyserades sedan i programmet Open Access. Protokollet för Realtids-PCR som skapades i Open Access programmet var samma som för ABI, men med kortare tider. Protokollet var uppvärmning på 95˚C i 2 min följt av 45 cykler av 95˚C i 8 sek och 60˚C i 25 sek. Proverna sattes i rack som sedan placerades i Panther Fusion efter att PPR-mix och PCR cartridge placerats på sina respektive platser.
Vissa prov, som primärt blev negativa, testades om utan IK-primers med Panther fusion för att undersöka om känsligheten kunde förbättras. Slutgiltigt resultat inkluderade även Panther fusion analyserna som utfördes utan IK-primers.
Statistik
För att jämföra fördelning av resultat mellan metoderna användes Fishers exakta test (https://www.graphpad.com/quickcalcs/contingency1). Signifikant testning utfördes på 0,05-nivån.
6
RESULTAT
MgCl2 optimering
Optimeringen av MgCl2-koncentrationen utfördes endast för Panther Fusion. MgCl2-koncentrationen var mest optimal vid 3mM eftersom både vildtyp (CCUG 57601) och mutant NG stam (GC 106 RES) gav amplifieringskurvor med rätt prober, och att vildtypstammen gav mutant-signal som var under RFU 200 som var cut off (Figur 1). Sålunda bestämdes att 3 mM MgCl2 skulle användas till Panther Fusion metoden.
Figur 1.Vildtyp (WT) NG och mutant (Mut) NG som PCR-testades för påvisning av ciprofloxacin-känslighet och resistens med olika MgCl2-koncentrationer med Panther Fusion.
Längst till höger visas förklaring till vad respektive kurva representerar. WT 4 FAM = [vildtyp NG, 4mM MgCl2, FAM detektorsignal hos prob]. Mut 4 VIC = [Mutant NG, 4mM MgCl2, VIC
detektorsignal hos prob].
Testning av prober
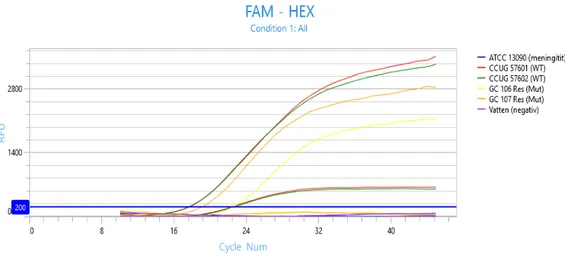
Utvidgad testning av olika stammar med PCR för att undersöka om proberna var specifika med Panther Fusion. Analysen visade att proberna gav korrekt signal med Panther Fusion och metoden var specifik för NG då Neisseria meningitidis (ATCC 13090) blev negativ, som visades i figur 2.
Figur 2. Vildtyp (WT) NG och mutant (Mut) NG stammar som PCR-testats för påvisning av
ciprofloxacin-känslighet och resistens resulterade i signal med korrekt prob med Panther Fusion. Kurvor som går över den blå linjen är positiva. Stammar som CCUG 57601 och CCUG 57602 som har två kurvor över den blå linjen, en för WT och en för Mut-proben(låg kurva nära blå linjen), anses vara WT då den har mycket starkare signal för WT än Mut.
7
Känslighetsanalys Panther Fusion
Resultatet av spädningsserier med sjunkande koncentration av positiva
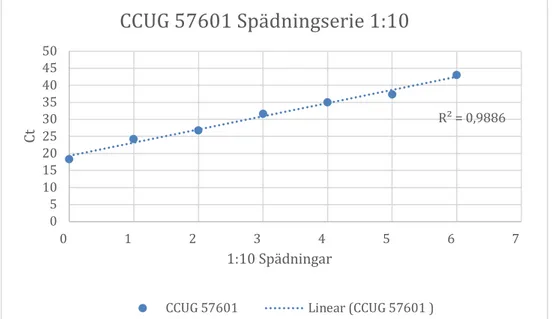
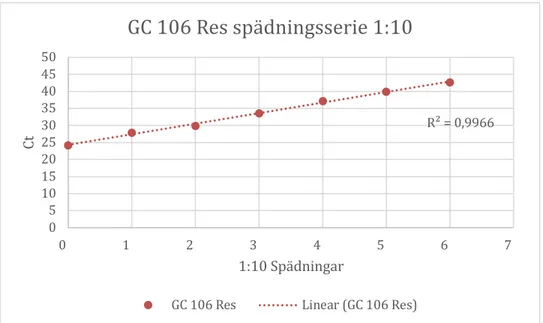
kontrollerna CCUG 57601, som var ciprofloxacin-känslig, och GC 106 Res, som var ciprofloxacin-resistent, NG stammarna visade att positiv signal genererades ner till spädning 10-6. Resultaten visas i tabell 5 och figurerna 3 och 4.
Tabell 5. 1:10 spädningsserie från 0 till 6 för CCUG 57601 och GC 106 Res där 0 spädningen är
ospädd lösning. CT=”Cykel of treshold” är det cykelvärde där kurvan når tröskelvärdet för PCR.
Spädning Log-10-faktor Ciprofloxacin-känslig NG CT (CCUG 57601) Ciprofloxacin- resistent NG CT (GC 106 Res) 0 18,4 24,2 1 24,3 27,9 2 26,8 29,9 3 31,6 33,6 4 35 37,2 5 37,3 39,9 6 43,1 42,7
Figur 3. 1:10 spädningsserie från 0 till 6 för CCUG 57601, Ciprofloxacin-känslig NG stam, som
visade ett R2-värde på 0,9886.
R² = 0,9886 0 5 10 15 20 25 30 35 40 45 50 0 1 2 3 4 5 6 7 Ct 1:10 Spädningar
CCUG 57601 Spädningserie 1:10
CCUG 57601 Linear (CCUG 57601 )8
Figur 4. 1:10 spädningsserie från 0 till 6 för GC 106 Res, Ciprofloxacin-resistent NG stam, som
visade ett R2-värde på 0,9966.
Patientprov
Femtio patientprov analyserades både med ABI Realtids-PCR och Panther Fusion. Proverna som reagerade med proben för vildtyp betecknas WT, proverna som reagerade med proben för den muterade sekvensen betecknas Mut och proverna som inte fick någon reaktion betecknas 0.
ABI
Sammanställningen av resultaten från ABI visas i tabell 6. Sammanställningen visade att 90 %, 45 av 50, av proverna resulterade i positiv signal. Resistens påvisades hos 62%, 28 av 45 av de prover där ABI kunde detektera genen, vilket visas i tabell 6. Resultaten för individuella prov med ABI visas i Bilaga, tabell 1.
Tabell 6. Sammanställning av resultaten från ABI. WT är Ciprofloxacin-känsliga prover, MUT är
Ciprofloxacin-känsliga prover och 0 är prover som inte fick något resultat.
ABI Resultat ANTAL WT 17 MUT 28
0 5
Panther Fusion
Resultaten från olika analys-omgångar från Panther Fusion Open Access visas i figurerna 6–9 och tabell 5. Resultaten från patient-prover visas i tabell 6 och 7 samt figurerna 6–9 där figurerna visar resultat för WT- och MUT-proberna. Den horisontella linjen i figurerna visar gränsen för positivt prov.
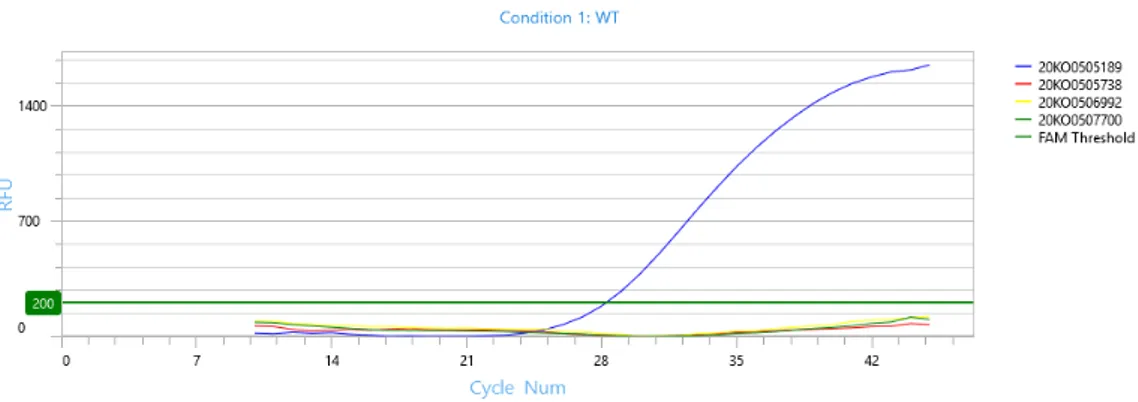
Fjorton prov resulterade i signal för WT NG, och 3 representativa prov visas i Figur 6. R² = 0,9966 0 5 10 15 20 25 30 35 40 45 50 0 1 2 3 4 5 6 7 Ct 1:10 Spädningar
GC 106 Res spädningsserie 1:10
GC 106 Res Linear (GC 106 Res)9
Figur 6. Representativa prover som reagerade med WT-proben. CCUG 57601 var positiv kontroll
för WT. Grön linje visar gräns för positivitet.
Tjugosex prov resulterade i signal för Mut NG, och 6 representativa prov visas i Figur 7.
Figur 7. Representativa prover som reagerade med Mut-proben. GC 106 Res var positiv kontroll
för Mut.
För att undersöka om känsligheten kunde ökas med ny PPR-mix utan primers för intern kontroll testades 8 prov som tidigare blivit negativa. Som positiva
kontroller användes patientprov (WT: 20KO0505189 och MUT: 20KO0504956). Endast positiva kontrollen blev positiv för WT, visas i figur 8, medans 3 av dessa 8 prov gav signal för Mut-prob, visas i figur 9.
Figur 8. Representativa prov som analyserades med WT-prob när PPR-mixen inte hade någon
10
Figur 9.Prover som analyserades med Mut-proberna när PPR-mixen inte hade någon intern kontroll. Kontroll för Mut var prov 20KO0504956 (röd).
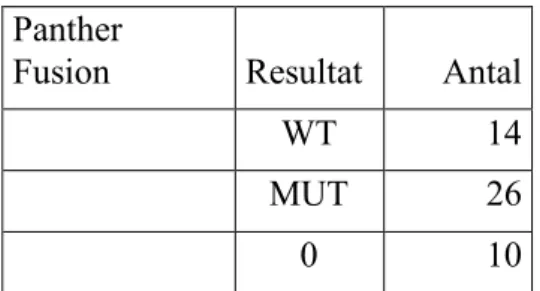
Totalt kunde genen påvisas i 80 %, 40 av 50, av proverna med Panther Fusion. Resistens påvisades hos 65%, 26 av 40, av de Panther Fusion-positiva proverna, visas i tabell 7. Resultat för individuella prov i Panther Fusion visas i bilaga tabell 2.
Tabell 7. En sammanställning av resultaten från Panther Fusion. WT är Ciprofloxacin-känsliga
prover, MUT är Ciprofloxacin-känsliga prover och 0 är prover som inte fick något resultat.
Panther
Fusion Resultat Antal
WT 14
MUT 26
0 10
En jämförelse mellan metoderna visade 90% (45/50) överensstämmelse av positiva och negativa resultat mellan Panther Fusion och ABI, vilket visas i tabell 8. Jämförelsen visade att ABI fick 5 fler positiva resultat än Panther Fusion.
Tabell 8. Jämför antalet prov där genen kunde påvisas med Panther Fusion gentemot ABI.
Panther + - ABI + 40 5 - 0 5 Odling
Proverna hade resistensodlats tidigare, handledare tillhandahöll resultaten av dessa odlingar. Resultaten för odlingarna enligt handledare visas i tabell 9. Sammanställningen visar att av 49 odlingar fick 24, 49 % av odlingarna, ett resultat. 0 indikerar att bakterierna inte kunde odlas. R betyder att bakterierna var resistenta och S att bakterierna var känsliga mot antibiotika. Resultat av odlingar för individuella prov visas i bilaga tabell 3.
11
Tabell 9. En sammanställning av resultaten från odlingarna. S är prover som är
Ciprofloxacin-känsliga, R är prover som är Ciprofloxacin-resistenta och 0 är prover som inte fick något resultat vid odling.
Odlingar Resultat Antal
S 8
R 16
0 25
En jämförelse av resultaten från de två PCR-metoderna visade att båda hade en perfekt överenstämmelse med odlingarnas resultat när det gäller vilka prov som var känsliga eller resistenta, där både PCR och odling fått resultat som visade resistenta och känsliga NG-isolat för Ciprofloxacin. ABI hade perfekt
överenstämmelse med 15 resistenta och 8 känsliga odlade NG-isolat. Panther Fusion hade perfekt överenstämmelse med 14 resistenta och 8 känsliga odlade NG-isolat.
Statistik
ABI-metoden påvisade ciprofloxacin-resistens eller känslighet hos 90% (45/50) av NG-positiva prover (APTIMA COMBO2), jämfört med 49% (24/49) hos parallellt tagna prov vilka odlats traditionellt för resistensbestämning (P<0,0001). Panther Fusion-metoden påvisade ciprofloxacin-resistens eller känslighet hos 80% (40/50) av NG-positiva prover (APTIMA COMBO2), jämfört med 49% (24/49) hos parallellt tagna prov vilka odlats för resistensbestämning (P=0,0016).
DISKUSSION
Ett intressant projekt som försöker hitta ett mer effektivt sätt att undersöka resistens mot Ciprofloxacin-antibiotika hos NG. Eftersom funktionen Open Access på Panther Fusion instrumentet inte tidigare har använts vid klinisk mikrobiologiska laboratoriet utfördes flera olika analyser med denna tekniken jämfört med den mer beprövade ABI-metoden.
För att undersöka analysens känslighet vid användningen av Panther Fusion iordningsställdes en spädningsserie. Dock kunde spädningsanalysen inte ange den exakta analytiska känsligheten, det vill säga det minsta antalet bakterier som analysen kunde påvisa, eftersom antalet bakterier i urspungslösningen inte hade bestämts.
Det rekommenderas från tillverkaren av Panther Fusion att bestämma optimal MgCl2-koncentration för sin PCR. MgCl2-koncentrationer som testades var 2mMm, 3mM och 4 mM. MgCl2 koncentrationen hos ABI PCR standardmix anges inte av tillverkaren men långvarig användning av PCR-mixen till olika PCR-analyser i laboratoriet har visat standardmixen fungerar tillfredställande utan att optimering av MgCl2 koncentrationen görs. Beträffande känsligheten hos Panther Fusion så konstaterades att känsligheten förbättrades något genom att utesluta primerna för interna kontrollen (Figur 8 och 9). Den förbättrade känsligheten kan kanske bero på att amplifieringen av gyrA blir något mer effektiv om samtidig amplifiering av kontrollen inte sker. Det bestämdes att 50 prover var tillräckligt med prov för detta projekt. Patientproverna analyserades
12
dock i grupper om färre prov då det var viktigt att säkerställa att metoderna visade reproducerbarhet, tex för positiva och negativa kontroller.
MgCl2 optimeringen visade att en koncentration på 3 mM var optimal då proverna blev klart positiva, till skillnad från 2 mM, och proverna blev inte positiva för både WT och Mut, till skillnad från 4 mM. Detta visas i figur 1.
Känslighetstestet resulterade i ett Ct-värde under 45 till en spädningsfaktor på 10-6, vilket indikerade att proverna kunde spädas upp till en miljon gånger och ändå resultera i ett positivt resultat. Detta indikerar också att analysen har en hög känslighet. Detta visas i figur 3–4 och tabell 5.
Tabell 8 visar att ABI genererade 5 fler positiva resultat än Panther Fusion. Denna skillnad kan bero på att instrumenten hade olika protokoll. ABI hade samma antal cykler som Panther Fusion; 45, men cyklerna hade mer tid för varje steg i ABI. Panther Fusion kunde inte ha längre cykler då programmet har i åtanke att så kallade kit-tester, standardiserade kit från Hologic, utförs samtidigt som Open Access labbtester utförs. Ett protokoll tillåts därför inte vara för långt, så att labbtesterna inte stör det vanliga arbetet. Det hade varit lättare att jämföra
instrumentens resultat om de följt samma protokoll, men som det ser ut just nu får ABI fler positiva resultat än Panther Fusion. Ändringar i protokoll kan göras i framtida tester för att se om resultaten blir mer lika. Dock har Panther fusion färre manuella steg än ABI då nukleinsyraextraktion utförs i instrumentet.
När analyserna jämförs, fick Panther fusion resultat för 40 av 50 prov (80%). ABI fick resultat för 45 av 50 (90%) och odling kunde resistensbestämma 24 av 49 (49,0%). Antalet prover var lågt, men resultaten ser lovande ut för att den molekylärbiologiska metoden är att föredra framför odling beträffande Ciprofloxacin-resistensbestämning hos NG.
KONKLUSION
Båda molekylärbiologiska metoderna ABI och Panther Fusion påvisade betydligt fler ciprofloxacin- resistenta och -känsliga NG-isolat än odling.
TACK
Tack till Hologic som utan kostnad bidrog med reagens och förbrukningsmaterial till analyserna med Panther Fusion.
13
REFERENSER
[1] FASS, (2019) Ciprofloxacin Mylan. >
https://www.fass.se/LIF/product?nplId=20000531000115&userType=0 <HTML (2020-02-02)
[2] CDC, (2018) Gonococcal Isolate Surveillance Project (GISP)
Profiles,2017.>https://www.cdc.gov/std/stats17/gisp2017/default.htm <HTML
(2020-02-02)
[3] Allan-Blitz L T, Wang X, Klausner J D, (2017) Wild-type Gyrase A Genotype of Neisseria gonorrhoeae Predicts In Vitro Susceptibility to Ciprofloxacin: A Systematic Review of the Literature and Meta-analysis. Sex transm dis, 44, 261– 265.
[4] Belland R J, Morrison S G, Ison C, Huang W M, (1994) Neisseria gonorrhoeae acquires mutations in analogous regions of gyrA and parC in fluoroquinolone-resistant isolates. Mol Microbiol, 14, 371–380.
[5] Unemo M, Del Rio C, Shafer W M, (2016) Antimicrobial Resistance
Expressed by Neisseria gonorrhoeae: A Major Public Health Problem in the 21st Century. Microbiol spectr, 4.
[6] Life Technologies Corporation, (2009) Applied Biosystems 7500 Fast and
7500 Real-Time PCR Systems.
>https://assets.thermofisher.com/TFS-Assets/LSG/brochures/cms_072493.pdf< PDF (2020-03-28)
[7] Hologic, (2020) The Panther Fusion System >https://pantherfusion.com/< HTML (2020-03-28)
[8] Hologic, (2020) Panther Fusion Open Access.
>https://www.hologic.com/hologic-products/diagnostic-solutions/panther-fusionr-open-accesstm< HTML (2020-03-28)
[9] Peterson S W, Martin I, Demczuk W, Bharat A, Hoang L, Wylie J, Allen V, Lefebvre B, Tyrrell G, Horsman G, Haldane D, Garceau R, Wong T, Mulvey M R, (2015) Molecular Assay for Detection of Ciprofloxacin Resistance in Neisseria gonorrhoeae Isolates from Cultures and Clinical Nucleic Acid Amplification Test Specimens. J Clin Microbiol, 53, 3606–8.
14
BILAGA
Tabeller. Fulla tabeller för alla prover med alla resultat från alla metoder, ABI,
Panther fusion respektive odling.
Tabell 1. Resultat för ABI. Resistenta prov kallas Mut, känsliga kallas WT och prov som inte fick
något resultat är 0.
Patient Provnummer Resultat av ciprores för ABI
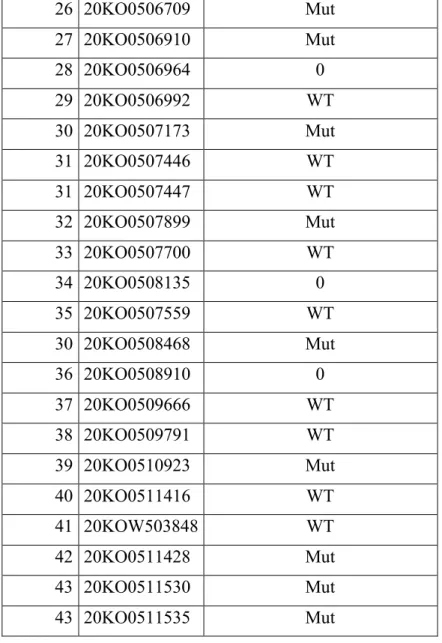
1 20KO0503810 WT 2 20KO0503438 WT 3 20KO0503331 WT 4 20KO0505189 WT 5 20KO0505176 WT 6 20KO0505746 WT 7 20KO0505536 WT 8 20KO0503715 Mut 9 20KO0504141 Mut 10 20KO0503517 Mut 11 20KO0502624 Mut 12 20KO0504361 Mut 13 20KO0504361 Mut 14 20KOW500594 Mut 15 20KO0505206 Mut 16 20KO0504956 Mut 17 20KO0505969 Mut 18 20KO0506172 Mut 17 19KOW530390 Mut 19 20KO0506570 Mut 19 20KO0504686 Mut 20 20KO0504075 Mut 21 20KO0505216 Mut 22 20KO0503267 0 23 20KO0503557 0 23 20KO0503555 0 24 20KO0505738 WT 22 20KO0506393 Mut 25 20KO0505204 Mut
15 26 20KO0506709 Mut 27 20KO0506910 Mut 28 20KO0506964 0 29 20KO0506992 WT 30 20KO0507173 Mut 31 20KO0507446 WT 31 20KO0507447 WT 32 20KO0507899 Mut 33 20KO0507700 WT 34 20KO0508135 0 35 20KO0507559 WT 30 20KO0508468 Mut 36 20KO0508910 0 37 20KO0509666 WT 38 20KO0509791 WT 39 20KO0510923 Mut 40 20KO0511416 WT 41 20KOW503848 WT 42 20KO0511428 Mut 43 20KO0511530 Mut 43 20KO0511535 Mut
Tabell 2. Resultaten av Panther Fusion Open Access. Resistenta prov kallas Mut, känsliga kallas
WT och prov som inte fick något resultat är 0.
Patient Provnummer Resultat av ciprores för Panther Fusion
1 20KO0503810 WT 2 20KO0503438 WT 3 20KO0503331 WT 4 20KO0505189 WT 5 20KO0505176 WT 6 20KO0505746 WT 7 20KO0505536 WT 8 20KO0503715 Mut 9 20KO0504141 Mut 10 20KO0503517 Mut
16 11 20KO0502624 Mut 12 20KO0504361 Mut 13 20KO0504361 Mut 14 20KOW500594 Mut 15 20KO0505206 Mut 16 20KO0504956 Mut 17 20KO0505969 Mut 18 20KO0506172 Mut 17 19KOW530390 Mut 19 20KO0506570 Mut 19 20KO0504686 Mut 20 20KO0504075 Mut 21 20KO0505216 Mut 22 20KO0503267 0 23 20KO0503557 0 23 20KO0503555 0 24 20KO0505738 0 22 20KO0506393 MUT 25 20KO0505204 0 26 20KO0506709 Mut 27 20KO0506910 Mut 28 20KO0506964 0 29 20KO0506992 0 30 20KO0507173 Mut 31 20KO0507446 WT 31 20KO0507447 WT 32 20KO0507899 MUT 33 20KO0507700 0 34 20KO0508135 0 35 20KO0507559 WT 30 20KO0508468 Mut 36 20KO0508910 0 37 20KO0509666 WT 38 20KO0509791 WT 39 20KO0510923 MUT
17 40 20KO0511416 WT 41 20KOW503848 WT 42 20KO0511428 0 43 20KO0511530 Mut 43 20KO0511535 Mut
Tabell 3. Resultaten av odling. Resistenta prov kallas R, känsliga kallas S och prov som inte fick
något resultat är 0.
Patient Provnummer Resultat av ciprores för odling
1 20KO0503810 0 2 20KO0503438 0 3 20KO0503331 0 4 20KO0505189 S 5 20KO0505176 S 6 20KO0505746 0 7 20KO0505536 0 8 20KO0503715 R 9 20KO0504141 0 10 20KO0503517 R 11 20KO0502624 R 12 20KO0504361 0 13 20KO0504361 0 14 20KOW500594 R 15 20KO0505206 R 16 20KO0504956 R 17 20KO0505969 0 18 20KO0506172 R 17 19KOW530390 0 19 20KO0506570 R 19 20KO0504686 R 20 20KO0504075 R 21 20KO0505216 0 22 20KO0503267 R 23 20KO0503557 0 23 20KO0503555 0
18 24 20KO0505738 0 22 20KO0506393 R 25 20KO0505204 R 26 20KO0506709 Ej odlad 27 20KO0506910 R 28 20KO0506964 0 29 20KO0506992 0 30 20KO0507173 0 31 20KO0507446 S 31 20KO0507447 S 32 20KO0507899 0 33 20KO0507700 0 34 20KO0508135 0 35 20KO0507559 0 30 20KO0508468 0 36 20KO0508910 0 37 20KO0509666 S 38 20KO0509791 S 39 20KO0510923 0 40 20KO0511416 S 41 20KOW503848 S 42 20KO0511428 0 43 20KO0511530 R 43 20KO0511535 R