Examensarbete Malmö Universitet
15 hp Hälsa och samhälle
Biomedicinska analytikerprogrammet 205 06 Malmö Januari-Mars 2020
JÄMFÖRELSE MELLAN
PREPARATIONSMETODER AV
POSITIV BLODODLING INFÖR
DIREKT IDENTIFIERING MED
MALDI-TOF MS
1
JÄMFÖRELSE MELLAN
PREPARATIONSMETODER AV
POSITIV BLODODLING INFÖR
DIREKT IDENTIFIERING MED
MALDI-TOF MS
REMAN MATTI
Matti, R. Jämförelse mellan preparationsmetoder av positiv blododling inför direkt
identifiering med MALDI-TOF MS Examensarbete i biomedicinsk laboratorievetenskap, 15
högskolepoäng. Malmö universitet: Fakulteten för hälsa och samhälle, institution för
Biomedicinsk vetenskap, 2020.
Sepsis (blodförgiftning) är ett livshotande tillstånd som innebär att bakterier eller svampar tar sig in i blodbanan. Det sker ofta genom njurarna, lungorna eller sår i skadad hud. Symptom innefattar feber och allmän sjukdomskänsla. Positiva blododlingar prepareras och analyseras med matrix assisted laser desorption ionisation time-of-flight masspektrometri (MALDI-TOF MS) där blandas preparerade prov med matrix och bestrålas sedan med en laser.
Bestrålningen medför att proteinerna i provet joniseras och rör sig mot en detektor.
Proteinerna detekteras som ett spektrum som i sin tur jämförs med ett referensspektrum av en känd bakterie/svamp som finns lagrat i en databas. Dessutom erhålls ett score-värde över hur väl provet liknar ett referensprov. Arbetets syfte var att se vilken provberedningsmetod som gav ett tillförlitligt resultat i MALDI-TOF och ett bra score-värde för identifiering av
bakterier till art-nivå på kortast tid. Preparationsmetoderna innefattar rening av blod från röda blodkroppar och andra partiklar som stör MALDI TOF-instrument. Metoderna jämfördes med nuvarande Saponinmetod. Två av metoderna (Ferroni och Huang) gav dåliga resultat och jämförelsen avbröts efter två försök. Ett kommersiellt kit Sepsityper extraktion gav bra resultat där 79 % av analyserna gav identifiering till art-nivå. Motsvarande resultat med saponin-metoden var 33 %. Slutsatsen är att Sepsityper-extraktion-metoden gav betydligt bättre resultat än nuvarande Saponin-metod. Metoden var robust, användarvänlig, snabb och gav bra resultat både för gramnegativa och grampositiva bakterier.
Nyckelord: Blododlingar, gramnegativa bakterier, grampositiva bakterier, MALDI-TOF MS,
2
COMPARISON OF PREPARATION
METHODS FOR DIRECT
IDENTIFICATION FROM POSITIVE
BLOOD CULTURES USING
MALDI-TOF MS
REMAN MATTI
Matti, R. Comparison of preparation methods for direct identification from positive blood cultures using MALDI-TOF MS. Degree project in Biomedical Science, 15 Credit points. Malmö University: Faculty of health and society, Department of Biomedical Science, 2020. Sepsis (blood poisoning) is a life-threatening condition caused by bacteria or fungi entering the bloodstream. Pathogens often again access through the kidneys, lungs or wounds in damaged skin. Symptoms include fever, chills and general feeling of illness. Positive blood cultures are prepared and analyzed with matrix assisted laser desorption ionization time-of-flight mass spectrometry (MALDI-TOF MS) prepared samples are mixed with matrix and then irradiated with laser beam. The laser beam will be directed to each position in the MALDI plate that causes the proteins in the sample to ionize and move towards a detector. The protein pattern is presented in a spectrum that is compared with a reference spectrum of known bacteria / fungi, stored in a database. In addition, a score value was obtained on how well the sample is aligned to the reference sample. The aim of this study was to compare three methods in order to achieve the best score value for identification to species level. The time factor was also important. The preparation methods include purification of blood removing red blood cells and other particles that interfere with MALDI analysis. The methods were compared with the current Saponinmethod. The results of two methods (Ferroni and Huang) were not satisfactory and further comparison was interrupted after two experiments. A commercial kit Sepsityper with extraction gave good results with 79 % identification to species level. Corresponding results for the current Saponin-method was 33 %. In conclusion, the Sepsityper-extraction-method was superior to the current Saponin-method. The method was robust, user-friendly, rapid and gave good results from both negative and gram-positive bacteria.
Key words: Blood cultures, gramnegative bacteria, grampositive bacteria, Maldi-TOF MS,
3
FÖRORD
Jag är oerhört tacksam till min handledare Ingegerd Sjögren för hennes hjälp, stöd och vägledning genom arbetets gång. Vill gärna rikta ett stort tack till verksamhetschefen Arne och enhetschefen Marie-Louise på klinisk mikrobiologi vid Hallandssjukhus i Halmstad för möjligheten att utföra mitt examensarbete i deras verksamhet. Ett jätte stort tack till min familj och min kille som var alltid på min sida, stöttade och uppmuntrade mig genom hela utbildningen och för hans tålamod med mitt konstiga humör under tiden. Vill gärna rikta ett speciellt tack till min vän Karin för hennes hjälp och stöd med språket sedan kom jag ny till Sverige. Vill också rikta ett varmt tack till landet Sverige som gav mig chansen och allt möjligt för att kunna få en sån härlig och underbar utbildning.
4
INNEHÅLLSFÖRTECKNING
BAKGRUND ... 5
Grampositiva och gramnegativa bakterier ... 5
Olika metoder för identifiering av bakterier ... 6
Virtuo och blododligsflaskor ... 6
MALDI-TOF MS... 6
SYFTE ... 8
MATERIAL OCH METOD ... 8
Preparation av blod med nuvarande Saponin-metoden ... 8
Preparation av blod med Ferroni-metoden ... 8
Preparation av blod med Huang-metoden ... 8
Preparation av blod med Sepsityper-pellet-metoden ... 9
Preparation av blod med Sepsityper-extraktion-metoden ... 9
ETISK BEDÖMNING ... 9
RESULTAT ... 9
DISKUSSION ... 11
REFERENSER ... 13
5
BAKGRUND
Sepsis (blodförgiftning) och septisk chock är ett livshotande tillstånd och innebär att
bakterierna eller svampar har tagit sig in i blodbanan. Det sker ofta genom njurarna, lungorna eller sår i skadad hud. Dess symptom innefattar feber och allmän sjukdomskänsla. Personer i alla åldrar kan drabbas av sepsis. Patienter med maligna sjukdomar, diabetes, alkoholism, granulocytopeni och svagt immunförsvar drabbas oftare än friska människor. I Sverige är E.
coli, S. aureus och S. pneumoniae de vanligaste infekterande mikroorganismer vid sepsis.
Vårdrelaterad sepsis orsakas oftast av bakterier ur familjerna Enterobacteriaceae,
Pseudomonas aeruginosa och enterokocker [1].
För det mesta utgår S. aureus infektionen från hudskador eller kateter. S. aureus angriper friska hjärtklaffar och kan ge upphov till endokardit. S.epidermidis (koagulasnegativa stafylokocker) ger upphov till kateterassocierad bakteriemi, speciellt bland patienter med hematologiska maligniteter och benmärgstransplantation. Streptocuccus pneumoniae orsakar pneumoni (lunginflammation) som i sin tur kan ge upphov till sepsis. E. coli kan spridas från njurarna till blodet och ger upphov till sepsis. E. coli kan också nå blodet direkt från tarmen vid trauma eller blödningar [1].
För att underlätta och förbättra diagnostisken vid sepsis görs blododlingar, vanligtvis två aeroba odlingar och två anaeroba odlingar i blododlingsflaskor. Den aeroba
blododlingsflaskan tas alltid först och den anaeroba flaskan sist. Orsaken till det är att det finns luft i slangarna som stör tillväxten av anaeroba bakterier. När det gäller barn görs oftast enbart en aerob odling eftersom barn sällan har bakteriemier med anaeroba bakterier. Nyfödda drabbas oftast av grupp B-streptokocker och E. coli. Små barn och ungdomar upp till 20-årsåldern drabbas vanligtvis av S. aureus, meningokocker och pneumokocker. Vid behandling av sepsis injiceras antibiotika direkt i blodet. Om sepsis upptäcks tidigt och diagnostiseras rätt blir patienten vanligtvis frisk. Vid allvarliga fall krävs intensivvård som uppehåller blodtryck, andning, blodcirkulation och njurfunktion [1].
Grampositiva och gramnegativa bakterier
Bakterier kan indelas i två huvudgrupper; grampositiva och gramnegativa bakterier baserat på cellväggens struktur. De vanligaste patogena grampositiva bakterier är stafylokocker och streptokocker vars patogena egenskaper kan förändras genom ökad-virulensfaktorer och ökad resistens mot antibiotika. Stafylokocker är runda grampositiva kocker och växer i klasar. Tio olika arter finns beskrivna hos människa så som S. aureus, S. epidermidis och S.
saprophyticus. Stafylokocker finns på huden på ett flertal människor utan att ge symptom och
kan orsaka olika infektioner såsom hud och sårinfektioner. Bakterierna är fakultativt anaeroba vilket innebär att de har förmåga att leva i både aeroba och anaeroba miljöer [2].
Utöver stafylokocker finns det även andra humanpatogena grampositiva kocker som
pneumokocker, beta-streptokocker och enterokocker. Streptokocker är grampositiva bakterier som växer i kedjor. De är oftast fakultativt anaeroba men vissa bakterier behöver mer syre och Gramnegativa bakterier dels in i kocker och stavar. Gramnegativa kocker; Neisseria och är
6
därmed aeroba bakterier. Streptokocker är katalasnegativa till skillnad från stafylokocker som är katalaspositiva. De delas in i alfa och beta streptokocker (streptokockus pyogenes) [2]. Gramnegativa bakterier dels in i kocker och stavar. Gramnegativa kocker; Neisseria och
Moraxella. Gramnegativa stavar; E. coli, Klebsiella pneumoniae, Proteus, Haemophilus influenzae, Salmonella, Shigella och Legionella. Den gramnegativa cellväggen är mycket
tunnare än grampositiva bakteriens cellvägg och innehåller peptidoglykaner. Dessutom har de gramnegativa bakterierna förutom ett cellmembran innanför cellväggen också ett yttre
cellmembran utanför cellväggen. Både det yttre och det inre cellmembranet består av två lager av fosfolipider men många av fosfolipiderna i det yttre cellmembranet är ersatta av
lipopolysackarid (LPS). LPS utgör en viktig faktor i virulensen hos gramnegativa bakterier [1].
Olika metoder för identifiering av bakterier
Nuvarande diagnostik på klinisk mikrobiologi på Hallands sjukhus i Halmstad baseras på direkt MALDI-TOF MS från blododlingar samt på gramfärgning av odlingar på agarplattor av MALDI-TOF positiva blododlingar. MALDI-TOF görs direkt från blododlingar innan de odlas ut på plattor. Gramfärgning ger ett primärt och snabbt svar. Gramfärgning identifierar bakteriens utseende och möjliggör tidig detektion av patogener. Gramfärgning är en viktig metod eftersom den ger ett snabb och säkert resultat så att patienten kan få korrekt behandling inom kort tid. Patientens överlevnadsmöjlighet ökas då markant. Trots att gramfärgning ger ett snabbt svar är den otillräcklig för att säkerställa korrekt bakterieart. I nuvarande läge vid klinisk mikrobiologi i Halmstad används en annan metod där positiva blododlingar odlas vidare på agarplattor under 6 timmar och bakteriekolonier som växer på agarplattorna analyseras med MALDI-TOF MS för art och familje-bestämma bakterierna. Den nuvarande preparationsmetoden för analys med MALDI-TOF från blododlingar (direkt MALDI-TOF) är väl fungerande men ger tillfälligtvis resultat som är svåra att tyda [3-4].
Virtuo och blododlingsflaskor
Blododlingsflaskorna inkuberas i en speciell inkubator en s.k. ”Virtuo” som är inställt på 36 C° och har 432 platser för blodflaskor. Inkubatorn består av en modul där blodflaskor
placeras. Inkubatorn vaggar blodflaskorna och avläser automatiskt deras bottnar flera gånger varje timme med en ljusdiod för att bestämma halten av CO2 i odlingsflaskan.
Odlingsflaskornas botten består av ett material som gulfärgas vid ökad mängd CO2. Flaskorna
inkuberas under 5 dagar. Vid tillväxt av bakterier larmas flaskan som positiv. Larmet orsakas av att bakterierna CO2 i blododlingsflaskan och det är CO2 produktion som registreras
(larmas). När en blododlingsflaska larmas som positiv beror detta på hur snabbt bakterierna växer. Exempelvis E. coli, som är en snabbväxande bakterie larmar efter 1-2 dagar medan anaeroba bakterier har längre tillväxthastighet kan larma först efter 5 dagar [1].
MALDI-TOF MS
Matrix-assisted laser desorption ionization-time of flight mass spectrometry (MALDI-TOF MS) är ett billigt, snabbt och pålitligt verktyg för identifiering av mikroorganismer som odlas på agarplattor eller i vätskor. MALDI-TOF MS är en analysmetod som kan artbestämma bakterier och svampar genom att analysera provens innehåll av proteiner. Olika bakterier och
7
svampar har egna och unika protein-profiler i ribosomerna som detekteras med MALDI-TOF MS. Sedan jämförs resultaten med kända mikroorganismers profiler som finns lagrade i en MALDI-biotyp-databas. Resultatet visas som ett score-värde som beräknas genom att analysresultaten jämförs med kända mikroorganismers data lagrade i databasen. Ett högt score-värde indikerar att mikroorganismen har identifierats [5-6].
Resultatet visar hur väl provet som utförs med MALDI-TOF matchar olika kända bakterier och svampar och en sannolik artbestämning kan göras. Artidentifiering görs från en koloni från primärplatta eller direkt från mediet i blododlingsflaskan. På en MALDI-platta stryks bakterier eller medium ut med en tandpetare som en tunn hinna på två positioner (dubbelprov) i MALDI-plattan. En MALDI-platta är gjord av metall och innehåller 96 ringar (positioner). Identifiering kan misslyckas om för mycket eller för lite material används. Efter utstrykning av provet på plattan täcks proven med en organisk lösning som kallas för matrixlösning [5,7]. Matrixlösning består av HCCA (α-cyano-4-hydroxycinnamic acid, C10H7NO3) som öppnar upp bakterierna så att proteiner blir fria. Positionerna med bakterier måste torkas innan de placeras i Maldi-TOF-instrument, matrixen och provet kristalliseras vid torkning. När allt torkas placeras plattan i Maldi-TOF-instrument. En laserstråle riktas mot varje position i Maldi-platta, vilket medför att provet joniseras. Provet detekteras och mäts sedan genom att olika mass-analyser används. I mikrobiologiska sammanhang används oftast time of flight analyser, dvs. proteinerna accelereras i ett vacuumrör (flight tube) mot detektorn. Hastigheten på joniserande proteinerna definieras som tiden det tar för proteinerna att passeras genom hela vacuumröret. Hastigheten är proportionell mot proteinets massa [5,7].
Små proteiner rör sig snabbt och stora rör sig långsamt i vacuumröret. Alla mikroorganismer lämnar ett s.k. molekylärt fingeravtryck på själva detektorn som i sin tur avslöjar proteinets massa och även talar om vilken bakterie innehåller provet. Proteinerna som detekteras visas i ett spektrum. Det finns karaktäristiska spektrum i en databas för olika bakteriearter och
familjer som det okända provet kan jämföras med. Maldi-TOF kan tidigt identifiera bakterier i blododlingar, vid urinvägsinfektioner och vid luftvägsinfektioner m.m. Resultatet visas som score-värde för hur bra provets spektrum stämmer överens med referensspektrum från en känd bakterie/svamp [5,7-8].
Vid analys med MALDI-TOF MS ligger score-värde mellan 0,000-3,000. Score-värde mellan 0,000-1,699 anger osannolik identifiering, score-värde mellan 1,700-1,999 anger en trolig identifiering, score-värde mellan 2,000-2,299 anger en tillförlitlig artidentifiering och ett värde mellan 2,300-3,000 anger en mycket tillförlitlig artidentifiering. Vid identifiering av positiva blododlingar med MALDI-TOF utnyttjades 1,700 som lägsta godkända score [7]. Score-värde mellan 1,700-1,999 indikerar till familje-identifiering medan score-värde över 2,000 indikerar till artidentifiering [9].
8
SYFTE
Syftet med projektet var att jämföra olika metoder för provpreparation från positiva blododlingar inför analys med MALDI-TOF MS. Samt tidsåtgången för varje
preparationsmetod för att se vilken metod som ger ett tillförlitligt och snabbt preliminärt svar på bakterieinnehållet i en blododling.
MATERIAL OCH METOD
I detta projekt användes Virtuo (Biomérieux), TOF-instrument (Bruker), MALDI-platta (Bruker), scan speed mini, centrifug (Thermo electron corporation), vortex (KEBO-lab).
Kemikalier: HPLC-grade water (Honeywell, Germany), 99,5 % etanol (CCS Healthcare AB), 96 % absolut etanol (CCS Healthcare AB), 5 % Saponinlösning (Sigma aldrich), 10 %
trifluoroacetic acid (Sigma aldrich), 70 % myrsyra (Honeywell, Germany), acitonitrile (Fisher Scientific), Sepsityper-kit (Bruker) och matrix.
Biologiskt material: 45 blododlingar som erhölls vid klinisk mikrobiologi i Halmstad.
Preparation av blod med nuvarande Saponinmetoden
Metoden utfördes genom att 1 ml blod blandades med 200 µl saponin-lösning i ett
eppendorfrör. Saponin bryter ner de röda blodkropparna. Röret blandades noggrant med en Vortex och inkuberades 5 minuter i rumstemperatur. Efter inkuberingstiden centrifugerades röret 1 minut i maxhastighet (16300 x g). Supernatanten avlägsnades och pelleten löstes upp i 1 ml dubbeldestillerat vatten och blandades med Vortex. Röret centrifugerades igen i 1 minut i maxhastighet och supernatanten avlägsnades. Pelleten torkades 2 minuter i rumstemperatur. Därefter applicerades provet (pelleten) i två positioner på MALDI-platta med hjälp av en tandpetare och lufttorkades. De applicerade proven belades med 1 µl 70 % myrsyra och när myrsyra hade torkat täcktes proven med 1 µl matrixlösning som fick lufttorkas [9-10].
Preparation av blod med Ferroni metoden
Metoden utfördes genom att 1 ml blod blandades med 200 µl saponin-lösning i ett
eppendorfrör. Röret inkuberades 5 minuter i rumstemperatur och därefter tillsattes destillerat vatten upp till 1,5 ml. Röret centrifugerades 16600 x g i 1 minut och supernatanten
avlägsnades. Sedan tillsattes 30 µl 10 % trifluoroacetic acid till pelleten. Därefter pipetterades 1 µl av supernatanten i en prov-position på MALDI-platta och lufttorkades. Efter att provet hade torkat pipetterades först 1 µl 96 % absolut etanol och därefter 1 µl matrixlösning på provet [11].
Preparation av blod med Huang metoden
Metoden utfördes genom att 150 µl blod blandades med 30 µl saponin-lösning i ett
eppendorfrör. Röret inkuberades 8 minuter vid 35 °C. Därefter tillsattes 1 ml destillerat vatten till röret och provet inkuberades ytterligare 3 minuter vid 35 °C. Röret centrifugerades 50 x g
9
i 1 minut och supernatanten överfördes till ett nytt eppendorfrör. Det nya röret
centrifugerades 16200 x g i 1 minut varefter supernatanten avlägsnades och 1 ml destillerat vatten tillsattes till pelleten. Röret centrifugerades åter 16200 x g i 1 minut och supernatanten avlägsnades. Pelleten löstes i 15 µl 70 % myrsyra och 15 µl acetonitril och provet
centrifugerades 16200 x g i 1 minut. Därefter pipetterades 1 µl av supernatanten till en prov-position på MALDI-plattan, lufttorkades och belades med matrixlösning [12].
Preparation av blod med Sepsityper pellet metod
Metoden utfördes genom att 1 ml blod blandades med 200 µl lyseringsbuffert (Bruker) i ett eppendorfrör. Röret centrifugerades 16300 x g i 2 minuter och supernatanten avlägsnades. Därefter tillsattes 1 ml tvättbuffert (Bruker) till pelleten och röret centrifugerades 16300 x g i 1 minut. Supernatanten avlägsnades och därefter applicerades pelleten på två prov-positioner på MALDI-platta med hjälp av en tandpetare och fick lufttorka. En prov-position belades med 1 µl matrixlösning och den andra prov-positionen täcktes med 70 % myrsyra och belades därefter med matrixlösning [13].
Preparation av blod med Sepsityper-extraktion-metoden
Efter preparation av prov enligt ovan resuspenderades provet i 300 µl dubbeldestillerat vatten. Därefter tillsattes 900 µl 99,5 % etanol och röret blandades med Vortex i 10 sekunder. Röret centrifugerades 16 300 g i 2 minuter och därefter avlägsnades supernatanten. Röret
centrifugerades åter 16300 x g i 2 minuter för att resterande etanol skulle kunna avlägsnas. Pelleten löstes i 2-50 µl 70 % myrsyra. Val av volym myrsyra berodde på pelletens storlek. Lika volym acetonitril tillsattes till röret och provet centrifugerades 16300 x g i 2 minuter. Därefter pipetterades 1 µl av supernatanten till två prov-positioner på MALDI-platta och fick lufttorka. Till sist belades proven med matrixlösning och fick lufttorka [13].
ETISK BEDÖMNING
Projektet krävde inte någon etisk bedömning eftersom projektet utfördes för att bestämma bakterier som ändå skulle ha artbestämts från blododlingar vid klinisk mikrobiologi,
Hallandssjukhus, Halmstad. Inga personliga uppgifter som namn eller personnummer kunde kopplas till resultatet. Projektet innebar inga fysiska eller psykiska ingrepp på nu levande eller avliden människa.
RESULTAT
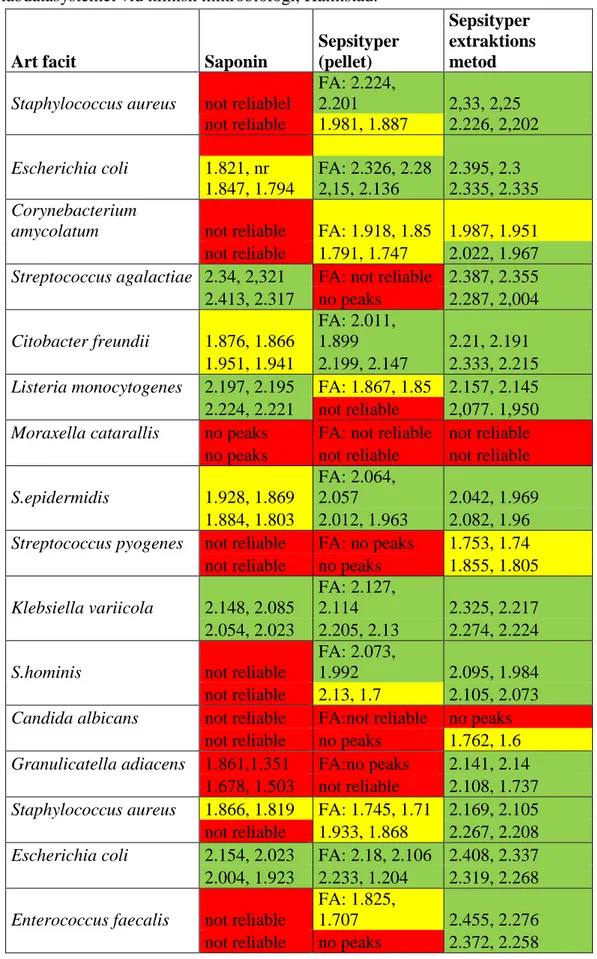
Resultaten utgjordes av score-värden från MALDI-TOF MS. Score < 1,699 (röd) innebar att ingen identifiering av mikroorganismer kunde göras, ett score-värde mellan 1,700-1,999 (gul) visade på en trolig identifiering till familje-nivå, score-värde mellan 2,000-2,299 (grön) visade en tillförlitlig identifiering av mikroorganismen ner till art-nivå. Detaljerade resultat
10
från MALDI-TOF MS analyser av prov från blododlingar preparerade med de olika provberedningsmetoderna som undersöktes visas i bilaga 1.
Med Ferroni-metoden preparerades prov från 11 blododlingar innehållande ett ganska stort antal olika bakteriestammar men resultaten visade att inga mikroorganismer kunde
identifieras i proven med MALDI-TOF MS. För att säkerställa de negativa resultaten
upprepades analyserna av en del av proven och dessa analyser blev också negativa (se bilaga 1). Tidsåtgången för metoden var ca 20 minuter.
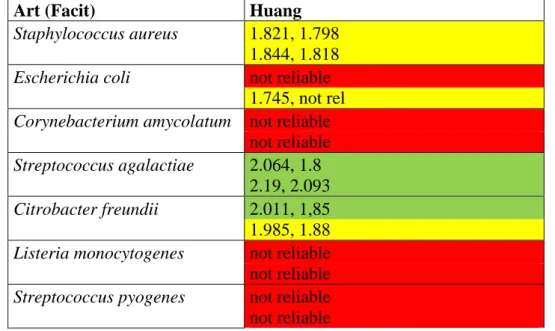
Med Huang-metod analyserades 7 blododlingar innehållande olika bakterier. Staphylococcus
aureus, Escherichia coli, Streptococcus agalactiae samt Citrobacter freundii kunde
identifieras i proven med score-värden mellan 1,800-2,200. Medan Corynebacterium
amycolatum, Listeria monocytogenes och Streptococcus pyogenes inte kunde identifieras (se
bilaga 1). Tidsåtgången för metoden var ca 30 minuter.
pellet utan myrsyra metod var likvärdig Saponin-metoden (se figur 1). Sepsityper-pellet med myrsyra metod gav viss förbättring, men Sepsityper-extraktion-metod gav bättre resultat och var mer användarvänlig.
Figur1: Sammanställning av procentuella resultat vid artbestämning med Maldi-TOF MS som
utfördes på positiva blododlingar preparerade med Saponin respektive Sepsityper metod. Ingen identifiering, score < 1,699, trolig identifiering, score-värde mellan 1,700-1,999 och tillförlitlig identifiering på art-nivå, score-värde mellan 2,000-2,299.
46 % 21 % 33 % Saponin-metod 33 % 27% 40 % Sepsityper-pellet-metod med myrsyra 9 % 12 % 79 % Sepsityper-extraktion-metod 49 % 21 % 30 % Sepsityper-pellet-metod utan myrsyra
11
Sepsityper preparationssmetoden (Sepsityper extraktion) gav bäst resultat med högst andel mikroorganismer identifierade på art-nivå. I proven identifierades 79 % av proven med korrekt mikroorganism (figur 1). Metoden var robust och snabb med en tidsåtgång på ca 20 minuter. Saponin-metoden som är preparationsmetoden, som idag används på klinisk
mikrobiologi, Hallandssjukhus, gav tillförlitliga identifikationer på art-nivå i endast 33 % av proven. Jämfört med detta är resultaten erhållna med Sepsityper-extraktion-metoden en klar förbättring.
Blododlingarna innehöll både grampositiva och gramnegativa bakterier, och en jästsvamp. Andelen grampositiva bakterier var 21st. Av dessa gav 17 st tillförlitliga resultat till art-nivå med Sepsityper-extraktion-metod. Andelen blododlingar som innehöll gramnegativa
bakterier var 11 st. Av dessa kunde 10 identifieras till art-nivå. En blododling innehöll jästsvamp, Candida albicans, och den identifierades till familje-nivå med Sepsityper-extraktion-metod.
DISKUSSION
Syftet med projektet var att jämföra olika metoder för provpreparation från positiva blododlingar inför identifiering av bakterier med MALDI-TOF MS. Samt tidsåtgången för varje preparationsmetod för att se vilken metod som ger ett tillförlitligt och snabbt preliminärt svar på bakterieinnehållet i en blododling. En tidig och effektiv behandling av patienter som drabbats av sepsis är avgörande för att minska dödligheten och sjukhusrelaterade kostnader. Det är därför viktigt att utveckla snabba diagnostiska metoder för identifiering av
mikroorganismer som orsakar sepsis och blodinfektioner [14]. MALDI-TOF MS är ett användbart instrument för snabb identifiering av bakterie/svamp-art i positiva blododlingar. För att exakta och tillförlitliga resultat skall erhållas måste provberedning göras inför analys med MALDI-TOF MS. Optimal provberedning måste göras för att minska antalet falska negativa så mycket som möjlig [3,9]. Resultatet från MALDI-TOF MS erhålls som ett score-värde och ett score-score-värde 0,000-1,699 orsakat av dålig kvalité på spektrumet från
massanalysen, får till följd att ingen artidentifiering av mikroorganismen kan göras [11]. Positiva blododlingar från patientprover där mikroorganismerna hade identifierats användes för provberedning inför analys med MALDI-TOF MS. Tre nya metoder för provpreparation; Ferroni-Huang- och Sepsityper-metoderna jämfördes med Saponin-metoden som är den nuvarande använda preparationsmetoden vid klinisk mikrobiologi på Hallandssjukhus. Med referensmetoden, Saponinmetoden, undersöktes 33 blododlingar positiva för mikroorganismer med olika bakteriearter. Metoden tog ca 15 minuter att utföra och gav otillräckligt eller
negativt värde i 46 % av blododlingarna, acceptabelt värde i 21 % av blododlingarna och i 33 % av blododlingarna kunde artbestämning ske. Metoden är fungerande men var ibland inte tillräckligt specifik för att kunna artbestämma mikroorganismen.
Med Ferroni-metoden kunde inte några mikroorganismer identifieras i något prov trots att prov preparerades från 11 blododlingar. Huang-metod användes för att preparera prov från 7 blododlingar med olika bakteriearter. Metoden gav ungefär samma andel negativa och
12
positiva prov som Saponin-metoden men metoden som sedan var opraktisk, innehöll många centrifugeringssteg, och tog längre tid ca 27-30 minuter än saponin-metod. Varken Ferroni- eller Huang-metoderna kan anses vara bättre än den på laboratoriet befintliga Saponin-metoden.
Metoden Sepsityper från Bruker undersöktes genom att prov preparerades från 33
blododlingar innehållande olika mikroorganismer. Provberedning inför analys med MALDI- TOF MS gjordes genom att erhållen pellet applicerades direkt, med och utan tillsats av myrsyra på analysplattan till MALDI- TOF MS instrumentet. Tillsättning av myrsyra gav bättre score-värde än utan myrsyra och tiden för provberedning var ca 15-17 minuter när pelleten applicerades direkt på instrumentets analysplatta och något längre, 23-25 minuter, när pellet löstes innan provet applicerades på instrumentets analysplatta. Med Sepsityper-pellet-metoden fick 67 % av proven acceptabelt värde eller kunde artbestämmas, score över 1,700 och bäst resultat med 79 % av proven som tillförlitlig identifiering på art-nivå var Sepsityper-extraktions-metoden.
Det finns ingen skillnad i tidsåtgången mellan Saponin och Sepsityper-pellet-metoderna. Båda tog ca 15-17 minuter att utföra. Skillnaden blir när det behövdes göras en extraktionsdel dvs. när den erhållna pelleten solubiliserades innan provet applicerades på instrumentets
analysplatta. Vid solubilisering (extraktion) av pelleten blev tidsåtgången något längre ca 23-25 minuter och resultatet blev mycket bättre. Fördelar med båda metoderna var att flera prover kunde prepareras samtidigt på ganska kort tid [3]. Den beräknade tidsåtgången inkluderar överföringen av provmaterialet till centrifugeringsröret (eppendorfröret) som gjordes med steril kanyl och spruta vilket tog 2-3 minuter. Båda metoderna var beroende av mängden mikroorganismer som fanns i provet.
Gramnegativa bakterier så som t.ex. E. coli är snabbväxande och i dess prover erhölls en stor mängd bakterier i alla prover som preparerades. Gramnegativa bakterier analyserades lättare med MALDI-TOF MS eftersom deras cellväggar är annorlunda uppbyggda än grampositiva bakteriers cellväggar. Enligt Loonen et al [3] kan Sepsityper-metoden bättre identifiera grampositiva bakterier jämfört med gramnegativa. Men denna studie visar inte någon skillnad vid identifiering av grampositiva respektive gramnegativa bakterier.
Studien visade att Sepsityper-metoden gav mer tillförlitliga resultat än Saponin-metoden. Sepsityper-extraktions-metoden gav trolig identifiering i 91 % av proven medan Saponin gav trolig identifiering i 54 % i proven. Sepsityper-metoden var robust, användarvänlig, snabb och gav bra resultat både för gramnegativa och grampositiva bakterier.
Sammanfattningsvis visar studien att Sepsityper-extraktions-metoden är ett pålitligt verktyg för direkt identifiering av mikroorganismer från positiva blododlingar. Detta resulterar i en snabb och säker artidentifiering. Saponin-metoden som används vid klinisk mikrobiologi, Hallandssjukhus måste förbättras eller bytas ut mot en effektivare metod såsom Sepsityper-extraktions-metoden.
13
REFERENSER
1. Brauner F, Arvidson K, Blomberg L, Castor TH (2015) Medicinsk mikrobiologi och
immunologi. Lund, Studentlitteratur AB.
2. Ericson E, Ericson T, Kan B (2018) Klinisk mikrobiologi, infektioner immunologi
vårdhygien. Stockholm, Sverige: Liber AB.
3. Loonen, A., Jansz, A., Stalpers, J., Wolffs, P. & Van Den Brule, A (2012) An
Evaluation of Three Processing Methods and the Effect of Reduced Culture Times for Faster Direct Identification of Pathogens From BacT/ALERT Blood Cultures by MALDI-TOF MS. Eur J Clin Microbiol Infect Dis, 31(7):1575-83
4. Bauman RW (2016) Microbiology with diseases by taxonomy. Pearson education limited.
5. Schubert, S. & Kostrzewa, M. (2017). MALDI-TOF MS in the Microbiology Laboratory: Current Trends. Curr Issues Mol Biol, 23:17-20
6. Lagace-Wiens, P. R., Adam, H. J., Karlowsky, J. A., Nichol, K. A., Pang, P. F.,
Guenther, J., et al. (2012) Identification of blood culture isolates directly from positive blood cultures by use of matrix-assisted laser desorption ionization-time of flight mass spectrometry and a commercial extraction system: analysis of performance, cost, and turnaround time. J. Clin. Microbiol, 50(10):3324-8
7. Patel R (2015) MALDI-TOF MS for the diagnosis of infectious diseases. Clinical
chemistry, 61(1):100-11
8. La Scola B, Raoult D (2009) Direct identification of bacteria in positive blood culture bottles by Matrix-Assisted Laser Desorption Ionization Time-of-Flight Mass
Spectrometry. Plos One, 25; 4 (11):e8041
9. Jung-Yien Chien J Y, Tai-Fen Lee, Shin-Hei Du, Shih-Hua Teng, Chun-Hsing Liao, Wang-Hui Sheng, Lee-Jene Teng, Po-Ren Hsueh (2016) Applicability of an in-house Saponin-based extraction method in Bruker biotypes Matrix-assisted laser
desorption/ionization Time of flight Mass spectrometry system for identification of bacterial and fungal species in positively flagged blood cultures. Front Microbiol, 7:1432
10. Chen J H, Ho P L, Kwan G S, She K K, Siu G K, Cheng V C, Yuen K Y, Yam W C (2013) Direct Bacterial Identification in Positive Blood Cultures by Use of Two Commercial Matrix-Assisted Laser Desorption Ionization–Time of Flight Mass Spectrometry Systems. J Clin Microbiol, 51(6):1733-9
14
11. Agne`s Ferroni, Ste´phanie Suarez, Jean-Luc Beretti, Brunhilde Dauphin, Emmanuelle Bille, Julie Meyer, Marie-Elisabeth Bougnoux, Alexandre Alanio, Patrick Berche, and Xavier Nassif1 (2010) Real-Time Identification of Bacteria and Candida Species in Positive Blood Culture Broths by Matrix-Assisted Laser Desorption Ionization–Time of Flight Mass Spectrometry. J Clin Microbiol, 48(5):1542-1548
12. Yong-Lu Huang, Qiao-Ling Sun, Jia-Ping Li, Yan-Yan Hu, Hong-Wei Zhou, Rong Zhang (2019) Evaluation of an in-house MALDI-TOF MS rapid diagnostic method for direct identification of micro-organisms from blood cultures. J Med Microbiol,
68(1):41-47
13. Munevver Kayin, Berivan Mert, Söhret Aydemir, Volkan Özenci (2019) Comparison of rapid BACpro® II, Sepsityper® kit and in-house preparation methods for direct identification of bacteria from blood cultures by MALDI-TOF MS with and without Sepsityper® module analysis. Eur J Clin Microbiol Infect Dis, 38(11):2133-2143 14. Osman Altun, Silvia Botero-Kleiven, Sarah Carlsson, Måns Ullberg, Volkan Özenci
(2015) Rapid identification of bacteria from positive blood culture bottles by MALDI-TOF MS following short-term incubation on solid media. Journal of medical
15
BILAGA 1
Tabell 1: Visar MALDI-TOF resultatet med Ferroni-metoden. I tabellen ses vilken art som
analyserades och MALDI-score som erhölls. Art-facit erhölls från labdatasystemet vid klinisk mikrobiologi, Halmstad.
Art (Facit) Ferroni
Escherichia coli no peaks x2
no peaks x2
Bacteroides fragilis no peaks x2
no peaks x2
Escherichia coli no peaks
Bacteroides fragilis no peaks
Staphylococcus aureus no peaks
Corynebacterium amycolatum no peaks Escherichia coli no peaks
Escherichia coli no peaks
Staphylococcus epidermidis no peaks
Escherichia coli no peaks
Bacteroides fragilis no peaks
Tabell 2: Visar MALDI-TOF resultatet med Huang-metoden. I tabellen ses vilken art som
analyserades och MALDI-score som erhölls. Art-facit erhölls från labdatasystemet vid klinisk mikrobiologi, Halmstad.
Art (Facit) Huang
Staphylococcus aureus 1.821, 1.798
1.844, 1.818
Escherichia coli not reliable
1.745, not rel
Corynebacterium amycolatum not reliable
not reliable
Streptococcus agalactiae 2.064, 1.8
2.19, 2.093
Citrobacter freundii 2.011, 1,85
1.985, 1.88
Listeria monocytogenes not reliable
not reliable
Streptococcus pyogenes not reliable
16
Tabell 3: Visar MALDI-TOF resultat vid jämförelse av saponin nuvarande metod och
sepsityper metod med dess två delar (pellet + extraktion). I tabellen ses vilken art som analyserades och score som erhölls vid respektive metod. Art-facit erhölls från
labdatasystemet vid klinisk mikrobiologi, Halmstad.
Art facit Saponin
Sepsityper (pellet)
Sepsityper extraktions metod
Staphylococcus aureus not reliablel
FA: 2.224,
2.201 2,33, 2,25
not reliable 1.981, 1.887 2.226, 2,202
Escherichia coli 1.821, nr FA: 2.326, 2.28 2.395, 2.3
1.847, 1.794 2,15, 2.136 2.335, 2.335
Corynebacterium
amycolatum not reliable FA: 1.918, 1.85 1.987, 1.951 not reliable 1.791, 1.747 2.022, 1.967
Streptococcus agalactiae 2.34, 2,321 FA: not reliable 2.387, 2.355 2.413, 2.317 no peaks 2.287, 2,004
Citobacter freundii 1.876, 1.866
FA: 2.011,
1.899 2.21, 2.191
1.951, 1.941 2.199, 2.147 2.333, 2.215
Listeria monocytogenes 2.197, 2.195 FA: 1.867, 1.85 2.157, 2.145 2.224, 2.221 not reliable 2,077. 1,950
Moraxella catarallis no peaks FA: not reliable not reliable
no peaks not reliable not reliable
S.epidermidis 1.928, 1.869
FA: 2.064,
2.057 2.042, 1.969
1.884, 1.803 2.012, 1.963 2.082, 1.96
Streptococcus pyogenes not reliable FA: no peaks 1.753, 1.74 not reliable no peaks 1.855, 1.805
Klebsiella variicola 2.148, 2.085
FA: 2.127,
2.114 2.325, 2.217
2.054, 2.023 2.205, 2.13 2.274, 2.224
S.hominis not reliable
FA: 2.073,
1.992 2.095, 1.984
not reliable 2.13, 1.7 2.105, 2.073
Candida albicans not reliable FA:not reliable no peaks not reliable no peaks 1.762, 1.6
Granulicatella adiacens 1.861,1.351 FA:no peaks 2.141, 2.14 1.678, 1.503 not reliable 2.108, 1.737
Staphylococcus aureus 1.866, 1.819 FA: 1.745, 1.71 2.169, 2.105 not reliable 1.933, 1.868 2.267, 2.208
Escherichia coli 2.154, 2.023 FA: 2.18, 2.106 2.408, 2.337 2.004, 1.923 2.233, 1.204 2.319, 2.268
Enterococcus faecalis not reliable
FA: 1.825,
1.707 2.455, 2.276
17
Staphylococcus
epidermidis not reliable FA: 1.847, 1.72 1.815, 1.723 not reliable 1.729, 1.709 1.829, 1.6
Parvimonas micra not reliable FA: not reliable not reliable not reliable not reliable not reliable
Escherichia coli 1.711, 1,665 FA: 2.042, 1.81 2.243, 2.092 not reliable not reliable 2.293, 2.095
Morganella morganii 2.233, 2.193
FA: 2.201,
2.177 2.494, 2.432
2.272, 2.25 2.033, 1.922 2.478, 2.404
Staphylococcus hominis not reliable
FA: 2.363,
2.327 2.226, 2.158
not reliable 2.214, 1.917 2.223, 2.154
Staphylococcus aureus not reliable not reliable 2.16, 2.138
not reliable no peaks 2.085, 2.014
Staphylococcus
epidermidis not reliable
FA: 1.717,
1.712 1.854, 1.792
not reliable not reliable 1.813, 1.7
Staphylococcus capitis 2.117, 2.086 FA: 2.189, 2.076 2.086, 2.065 1.905, 1.857 2.052, 1.981 1.991, 1.979 Klebsiella pneumoniae 2.215, 2.101 FA: 2.222, 2.157 2.362, 2.246 2.097, 1.989 2.088, 2.078 2.415, 2.337
Staphylococcus aureus 1.99, 1.989 FA: no peaks 2.435, 2.34
1.943, 1.918 no peaks 2.44, 2.321
Escherichia coli 2.061, 1.999
FA: 1.912,
1.906 2.158, 2.036
2.046, 2.013 1.952, 1.951 2.131, 2.092
Staphylococcus hominis no peaks FA: no peaks no peaks
not reliable no peaks no peaks
Staphylococcus
epidermidis not reliable FA: 1.973, 1.87 1.96, 1.755
not reliable not reliable 2.018, 1.96
Bacteroides
thetaiotaomicron 2.348, 2.343 FA: not reliable 2.265, 2.253
2.354, 2.352 not reliable 2.246, 2.18 Serratia marcescens 2.213, 2.165 FA: 2.248, 2.192 2.362, 2.333 2.253, 2.209 2.273, 2.25 2.291, 2.184 Bacillus cereus 2.233, 2.231 FA: 2.125, 2.096 2.328, 2.301 2.282, 2.212 not reliable 2.339, 2.295
Staphylococcus aureus 1.775, 1.748 FA: no peaks 2.043, 2.019